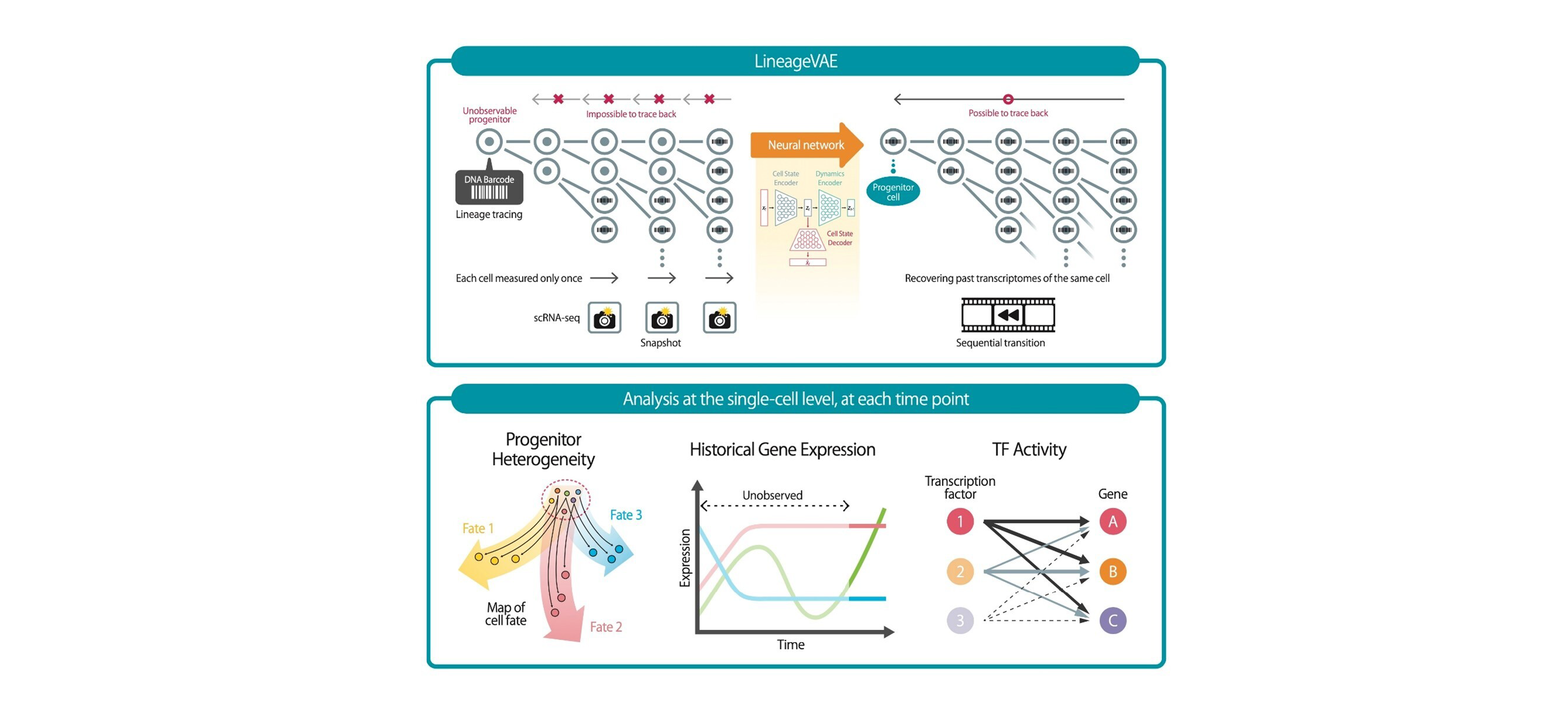
この手法は、scRNA-seqとLineage Tracing法を統合し、深層生成モデルの枠組みと同一バーコードを有する細胞系統は共通の祖先細胞をもつ姉妹関係にあるという性質を利用して、実験的には観測することができない、観測された細胞の祖先の細胞状態や過去の逐次的な遺伝子発現、転写因子による遺伝子制御ネットワークを一細胞解像度で分析することを可能にする技術です。本解析手法をマウス骨髄由来前駆細胞の造血現象、線維芽細胞から内胚葉前駆細胞へのダイレクトリプログラミング現象のデータに適用し、多数の細胞種や運命へと分岐する複雑な生命現象における観測されたそれぞれの細胞の祖先の細胞状態や、一細胞解像度で過去の遺伝子発現を通じた細胞状態遷移制御機構の推定が可能となりました。
細胞状態遷移を制御する機構の理解は、分化や発生、細胞応答、疾患、老化などの動的な生命現象の研究において不可欠です。LineageVAEは、scRNA-seqデータと Lineage Tracing データを用いて、それぞれの細胞の過去の状態遷移を復元し、一細胞解像度での共局在解析を実現することに成功しました。本解析手法は、細胞間相互作用の分子メカニズムに関する網羅的なデータに基づく仮説の提案を可能にし、再生医療の研究や新規の創薬標的の探索に役立つと期待されます。
本研究成果は、国際学術誌「Bioinformatics」に2024年8月22日にオンライン掲載されました。
※詳細につきましては、本取組に関するURLよりご覧ください。