「 深層生成モデルを活用した一細胞解像度での細胞状態遷移解析ツールの開発 」【 島村徹平 教授、小嶋泰弘 連携研究員】
公開日:2024.9.11
「 深層生成モデルを活用した一細胞解像度での細胞状態遷移解析ツールの開発 」
― 実験的に観測不可能なヒストリカルな遺伝子発現制御の解明へ ―
― 実験的に観測不可能なヒストリカルな遺伝子発現制御の解明へ ―

ポイント
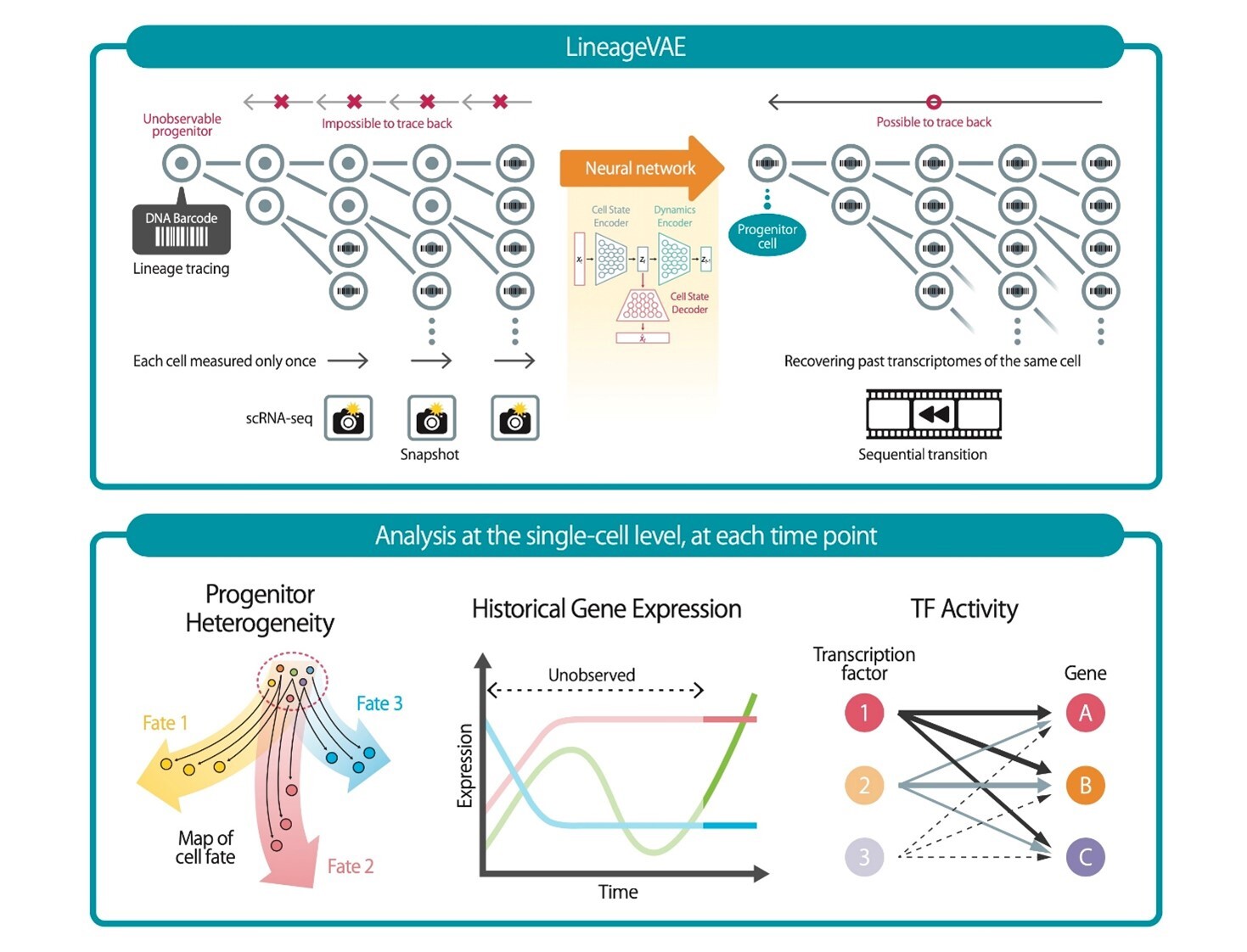
- シングルセルRNAシークエンス (scRNA-seq) ※1とLineage Tracing法 ※2を統合しヒストリカルな細胞状態の解析を行う手法LineageVAEを開発。
- 一細胞解像度での祖先の細胞状態や経時的な遺伝子発現、遺伝子制御ネットワークの推定が可能。
- 実験的に観測できない未分化細胞の細胞状態や経時的な遺伝子制御全体像の同定により、分化発生機構の解明や創薬標的の探索への応用に期待。
名古屋大学医学部医学科学生の間島滉一郎、国立がん研究センター研究所計算生命科学ユニットの小嶋泰弘独立ユニット長(東京医科歯科大学難治疾患研究所計算システム生物学分野連携研究員)、東京医科歯科大学難治疾患研究所計算システム生物学分野/名古屋大学大学院医学系研究科システム生物学分野の島村徹平教授らの研究グループは、生体組織内の細胞間共局在関係を解析するための画期的な情報解析手法「LineageVAE」を開発しました。
この手法は、scRNA-seqとLineage Tracing法を統合し、深層生成モデルの枠組みと同一バーコードを有する細胞系統は共通の祖先細胞をもつ姉妹関係にあるという性質を利用して、実験的には観測することができない、観測された細胞の祖先の細胞状態や過去の逐次的な遺伝子発現、転写因子による遺伝子制御ネットワークを一細胞解像度で分析することを可能にする技術です。本解析手法をマウス骨髄由来前駆細胞の造血現象、線維芽細胞から内胚葉前駆細胞へのダイレクトリプログラミング現象のデータに適用し、多数の細胞種や運命へと分岐する複雑な生命現象における観測されたそれぞれの細胞の祖先の細胞状態や、一細胞解像度で過去の遺伝子発現を通じた細胞状態遷移制御機構の推定が可能となりました。
細胞状態遷移を制御する機構の理解は、分化や発生、細胞応答、疾患、老化などの動的な生命現象の研究において不可欠です。LineageVAEは、scRNA-seqデータと Lineage Tracing データを用いて、それぞれの細胞の過去の状態遷移を復元し、一細胞解像度での共局在解析を実現することに成功しました。本解析手法は、細胞間相互作用の分子メカニズムに関する網羅的なデータに基づく仮説の提案を可能にし、再生医療の研究や新規の創薬標的の探索に役立つと期待されます。
本研究成果は、国際学術誌「Bioinformatics」に2024年8月22日にオンライン掲載されました。
この手法は、scRNA-seqとLineage Tracing法を統合し、深層生成モデルの枠組みと同一バーコードを有する細胞系統は共通の祖先細胞をもつ姉妹関係にあるという性質を利用して、実験的には観測することができない、観測された細胞の祖先の細胞状態や過去の逐次的な遺伝子発現、転写因子による遺伝子制御ネットワークを一細胞解像度で分析することを可能にする技術です。本解析手法をマウス骨髄由来前駆細胞の造血現象、線維芽細胞から内胚葉前駆細胞へのダイレクトリプログラミング現象のデータに適用し、多数の細胞種や運命へと分岐する複雑な生命現象における観測されたそれぞれの細胞の祖先の細胞状態や、一細胞解像度で過去の遺伝子発現を通じた細胞状態遷移制御機構の推定が可能となりました。
細胞状態遷移を制御する機構の理解は、分化や発生、細胞応答、疾患、老化などの動的な生命現象の研究において不可欠です。LineageVAEは、scRNA-seqデータと Lineage Tracing データを用いて、それぞれの細胞の過去の状態遷移を復元し、一細胞解像度での共局在解析を実現することに成功しました。本解析手法は、細胞間相互作用の分子メカニズムに関する網羅的なデータに基づく仮説の提案を可能にし、再生医療の研究や新規の創薬標的の探索に役立つと期待されます。
本研究成果は、国際学術誌「Bioinformatics」に2024年8月22日にオンライン掲載されました。
研究の背景
近年、scRNA-seqの技術の登場により、個々の細胞の遺伝子発現プロファイルを詳細に解析することが可能となり、生命現象や疾患における多様な細胞集団や遺伝子の関係への理解は大きく進展しました。しかし、scRNA-seqの解析は観測時に細胞を破壊するため、同一細胞における時系列的な遺伝子発現の変化や、分化・細胞分裂過程での動的な細胞状態の追跡ができないという課題があります。このため、従来の手法では、分化初期における祖先の細胞状態や生命現象の経時的な動態を捉えることが難しく、特に複数の細胞種に分化する生命現象の細胞状態変遷の方向性や祖先の細胞状態を正確に再構築することが困難でした。これらの課題に対処するために、DNA にバーコード配列を導入し細胞系譜を追跡するLineage Tracing法が開発され、動的な生命現象において異なる時刻における姉妹関係にある細胞の観測が可能となりましたが、依然として一細胞解像度で当該細胞の過去の遺伝子発現や祖先の細胞状態を直接観察することはできませんでした。
研究成果の概要
本研究では、一細胞解像度で過去その細胞がどのような状態を経て観測された細胞状態にたどり着いたのかを解明する新しい計算手法「LineageVAE」を開発しました。LineageVAEは、scRNA-seqとLineage Tracingを組み合わせ、細胞状態変遷のヒストリカルな経路や祖先の細胞状態を推定する深層生成モデル※3です。このモデルは、ニューラルネットワークを用いて、時間を遡る方向に細胞の潜在的な状態を推定します。さらにLineage Tracingデータを組み合わせることで、同一系統に属する細胞群が共有する祖先の細胞状態を推定します。これにより、観測されていない細胞状態を時間軸に沿って再構築することが可能になります。このアプローチにより、一細胞解像度で時間と共に変化する細胞状態およびヒストリカルな遺伝子発現、転写因子による遺伝子制御ネットワークを逐次的に推定することが可能になり、特定の細胞群の祖先の細胞状態や分化経路を高精度で推定することができます。本解析手法をマウス骨髄由来前駆細胞の造血現象、線維芽細胞から内胚葉前駆細胞へのダイレクトリプログラミングのデータに適用したところ、LineageVAEは、未観測の細胞状態や分化過程をマーカー遺伝子などの既知の知見と整合的に再構築できることが示されました。造血現象のデータセットではSerpinB1などの血球分化に重要な遺伝子を同定しました。また、ダイレクトリプログラミングのデータセットではリプログラミング成功後に高発現する遺伝子マーカーであるApoa1などについて既知の知見と時刻整合的な発現パターン復元しました。
研究成果の意義
本研究では、従来の実験手法および計算手法では困難だった祖先の細胞状態やヒストリカルな遺伝子発現を一細胞解像度で推定する革新的なアプローチであるLineageVAEを開発しました。この手法により、発生やがん、その他の疾患や障害のメカニズムなど、多岐にわたる動的な生命現象の時系列制御機構や遺伝子発現ダイナミクスをより深く理解することが可能になります。
LineageVAEは、細胞の分化の初期段階から観測された時刻に至るまでの過程を時間軸に沿って再構築できるため、新しい治療法の開発や、疾患の予防・治療における新たなターゲットの探索においても重要な役割を果たすと期待されます。特に、再生医療の分野では、初期の細胞状態や遺伝子発現の制御、分化経路の理解が進むことで、特定の細胞タイプへの分化を制御するための新たな戦略が提供され、組織再生や臓器移植の成功率を向上させる可能性があります。また、がん研究においても、がん細胞の起源に関与する遺伝子や進展経路を解明するための新たなアプローチとして期待されます。
これまでに一時刻においてのみ実験的に観測が可能であったオミクス解析において、祖先細胞の細胞状態まで時刻を遡りダイナミクスを捉えることにより、生命現象に対する理解が一層深まることが期待されます。この解析手法は、今後、様々な解像度の空間トランスクリプトームや広範な一細胞オミクスデータへとその応用範囲を広げる予定であり、これにより異なる分子レイヤーについての時空間情報を統合した組織間コミュニケーションの包括的な全体像の解明が可能となり、これは生物学及び医学分野における画期的な知見と治療法の発見に大いに貢献することでしょう。
LineageVAEは、細胞の分化の初期段階から観測された時刻に至るまでの過程を時間軸に沿って再構築できるため、新しい治療法の開発や、疾患の予防・治療における新たなターゲットの探索においても重要な役割を果たすと期待されます。特に、再生医療の分野では、初期の細胞状態や遺伝子発現の制御、分化経路の理解が進むことで、特定の細胞タイプへの分化を制御するための新たな戦略が提供され、組織再生や臓器移植の成功率を向上させる可能性があります。また、がん研究においても、がん細胞の起源に関与する遺伝子や進展経路を解明するための新たなアプローチとして期待されます。
これまでに一時刻においてのみ実験的に観測が可能であったオミクス解析において、祖先細胞の細胞状態まで時刻を遡りダイナミクスを捉えることにより、生命現象に対する理解が一層深まることが期待されます。この解析手法は、今後、様々な解像度の空間トランスクリプトームや広範な一細胞オミクスデータへとその応用範囲を広げる予定であり、これにより異なる分子レイヤーについての時空間情報を統合した組織間コミュニケーションの包括的な全体像の解明が可能となり、これは生物学及び医学分野における画期的な知見と治療法の発見に大いに貢献することでしょう。
用語解説
※1シングルセルRNAシークエンス (scRNA-seq)
一細胞ごとの区別をつけた状態でRNA分子の網羅的なシークエンシングを行うことにより、細胞ごとの遺伝子発現量の網羅的な定量化を行う手法。
※2Lineage Tracing法
母細胞の染色体に導入された DNA バーコード配列が娘細胞に受け継がれる性質を利用し、多細胞生物の発生過程など複雑な生命現象における各細胞系譜集団がどのような動態を示すかを追跡する手法。
※3深層生成モデル
深層学習によりデータの生成過程を記述した確率モデル。統計モデルの一種である生成モデルの学習にディープラーニングを用いた一連の手法の総称。様々な確立分布を組み合わせることで、 データや解析目的に応じた柔軟なモデルを設計することが可能。近年、一細胞トランスクリプトーム解析への応用が進んでいる。
一細胞ごとの区別をつけた状態でRNA分子の網羅的なシークエンシングを行うことにより、細胞ごとの遺伝子発現量の網羅的な定量化を行う手法。
※2Lineage Tracing法
母細胞の染色体に導入された DNA バーコード配列が娘細胞に受け継がれる性質を利用し、多細胞生物の発生過程など複雑な生命現象における各細胞系譜集団がどのような動態を示すかを追跡する手法。
※3深層生成モデル
深層学習によりデータの生成過程を記述した確率モデル。統計モデルの一種である生成モデルの学習にディープラーニングを用いた一連の手法の総称。様々な確立分布を組み合わせることで、 データや解析目的に応じた柔軟なモデルを設計することが可能。近年、一細胞トランスクリプトーム解析への応用が進んでいる。
研究助成
本研究は、日本学術振興会(JSPS)(課題番号20H04281、20K22839、22H04839、22H04925、23H04938c)、日本医療研究開発機構(AMED)(課題番号JP22ama221215、JP22ama221501、JP22ek0109488、JP23wm0325068、JP22wm0425007、JP23tm0424226)、科学技術振興機構(JST) ムーンショット型研究開発事業(目標2)(課題番号JPMJMS2025)および戦略的創造研究推進事業(課題番号JPMJAX20AB)、産業技術総合研究所(AIST) AI橋渡しクラウド(ABCI)、東京工業大学のスーパーコンピュータシステムTSUBAME、東京大学ヒトゲノム解析センターのスーパーコンピュータシステムSHIROKANE等の支援を受けて行いました。
論文情報
掲載誌:Bioinformatics
論文タイトル:LineageVAE: Reconstructing Historical Cell States and Transcriptomes toward Unobserved Progenitors
DOI:https://doi.org/10.1093/bioinformatics/btae520
研究者プロフィール

間島 滉一郎 (マジマ コウイチロウ) Majima Kouichiro
名古屋大学 医学部医学科
システム生物学分野 医学部6年生
・研究領域
システム生物学、バイオインフォマティクス、データ科学、人工知能
名古屋大学 医学部医学科
システム生物学分野 医学部6年生
・研究領域
システム生物学、バイオインフォマティクス、データ科学、人工知能

小嶋 泰弘 (コジマ ヤスヒロ) Kojima Yasuhiro
東京医科歯科大学 難治疾患研究所
計算システム生物学分野 連携研究員
国立がん研究センター研究所
計算生命科学ユニット 独立ユニット長
・研究領域
バイオインフォマティクス、データ科学、人工知能
東京医科歯科大学 難治疾患研究所
計算システム生物学分野 連携研究員
国立がん研究センター研究所
計算生命科学ユニット 独立ユニット長
・研究領域
バイオインフォマティクス、データ科学、人工知能

島村 徹平 (シマムラ テッペイ) Shimamura Teppei
東京医科歯科大学 難治疾患研究所
計算システム生物学分野 教授
名古屋大学 大学院医学系研究科
システム生物学分野 特任教授
・研究領域
システム生物学、バイオインフォマティクス、データ科学、人工知能
東京医科歯科大学 難治疾患研究所
計算システム生物学分野 教授
名古屋大学 大学院医学系研究科
システム生物学分野 特任教授
・研究領域
システム生物学、バイオインフォマティクス、データ科学、人工知能
問い合わせ先
<研究に関すること>
東京医科歯科大学 難治疾患研究所
計算システム生物学分野 島村 徹平(シマムラ テッペイ)
E-mail:shimamura.csb[@]tmd.ac.jp
名古屋大学 大学院医学系研究科
システム生物学分野 島村 徹平(シマムラ テッペイ)
E-mail:shimamura[@]med.nagoya-u.ac.jp
<報道に関すること>
東京医科歯科大学 総務部総務秘書課広報係
〒113-8510 東京都文京区湯島1-5-45
E-mail:kouhou.adm[@]tmd.ac.jp
名古屋大学医学部・医学系研究科 総務課総務係
〒466-8550 名古屋市昭和区鶴舞町65
E-mail:iga-sous[@]t.mail.nagoya-u.ac.jp
※E-mailは上記アドレス[@]の部分を@に変えてください。

