「タンパク質構造を変化させる遺伝子多型を同定する手法を開発」【高地雄太 教授】
公開日:2022.8.24
「タンパク質構造を変化させる遺伝子多型を同定する手法を開発」
― 選択的スプライシングの複雑性を読み解くことで疾患の病態解明に挑む ―
― 選択的スプライシングの複雑性を読み解くことで疾患の病態解明に挑む ―
ポイント
- タンパク質構造を変化させる選択的スプライシングに注目した、2つのスプライシングQTL解析※1の手法を開発しました。
- 無数のスプライシングイベントの中から、生物学的に意味のあるスプライシングアイソフォーム※2を効率良く検出することが可能となりました。
- この手法は、様々な疾患のゲノムワイド関連解析(GWAS)※3で明らかになった候補領域における病因メカニズムの解明に適応可能です。
研究の背景
遺伝子は、DNA配列からRNAが転写され、それを鋳型にタンパク質が翻訳されます。選択的スプライシングは、RNAの形成の段階で配列の取捨選択を行い、複数の成熟RNAを生み出しますが、タンパク質の多様性をもたらす最も重要なメカニズムです。この選択的スプライシングは、個人の遺伝子配列の違い(遺伝子多型)によって影響を受けることが知られており、そのような遺伝子多型をスプライシングQTLといいます。これらの一部は、免疫・アレルギー疾患、糖尿病などの様々な疾患の主要な病因メカニズムであることが知られています。しかし、選択的スプライシングはこれまでに想定されてきたよりも複雑なイベントであり、タンパク質構造を変えないものも含めると、ヒトゲノムにある約2万個の遺伝子から、数十万種以上に及ぶ成熟RNA(スプライシングアイソフォーム)を生み出すことがわかってきました。これらの中から生物学的に重要なスプライシングを同定し、疾患の病態解明に繋げるためには、何らかの戦略的な工夫が必要と考えられます。
研究成果の概要
スプライシングQTLによって影響をうけるスプライシングアイソフォームの大部分は、非翻訳領域(UTR; untranslated region)のみの変化であり、タンパク質に翻訳されるコーディング配列(CDS; coding sequence)には影響を与えない、すなわちタンパク質構造を変えないことが知られています。そこで研究グループは、同一のCDSを持つスプライシングアイソフォームを統合することで、タンパク質構造に変化を与えるスプライシングQTLのみを効率的に検出する手法、i2-rQTL解析(integrated-isoform ratio QTL解析, 図1A)を考案しました。この手法で、T細胞、B細胞などの5種類の免疫細胞で、スプライシングQTL解析を行ったところ、各細胞で300~600個のスプライシングQTLを同定することができました。
これらのi2-rQTL多型はタンパク質の構造変化を起こすことから、そのタンパク質の機能に大きく影響を与えることが考えられます。その結果、タンパク質が機能するパスウェイの下流の遺伝子群の発現に影響を与えると推定されます(図1B)。実際に、i2-rQTL多型は、タンパク質構造を変えないスプライシングQTL多型と比較して、他の遺伝子群の発現量により大きな影響を与えていることがわかりました(図1C)。また、これらのi2-rQTL多型の一部は、関節リウマチ、全身性エリテマトーデス、クローン病などの免疫疾患の原因となっていることも明らかになりました。
これらのi2-rQTL多型はタンパク質の構造変化を起こすことから、そのタンパク質の機能に大きく影響を与えることが考えられます。その結果、タンパク質が機能するパスウェイの下流の遺伝子群の発現に影響を与えると推定されます(図1B)。実際に、i2-rQTL多型は、タンパク質構造を変えないスプライシングQTL多型と比較して、他の遺伝子群の発現量により大きな影響を与えていることがわかりました(図1C)。また、これらのi2-rQTL多型の一部は、関節リウマチ、全身性エリテマトーデス、クローン病などの免疫疾患の原因となっていることも明らかになりました。
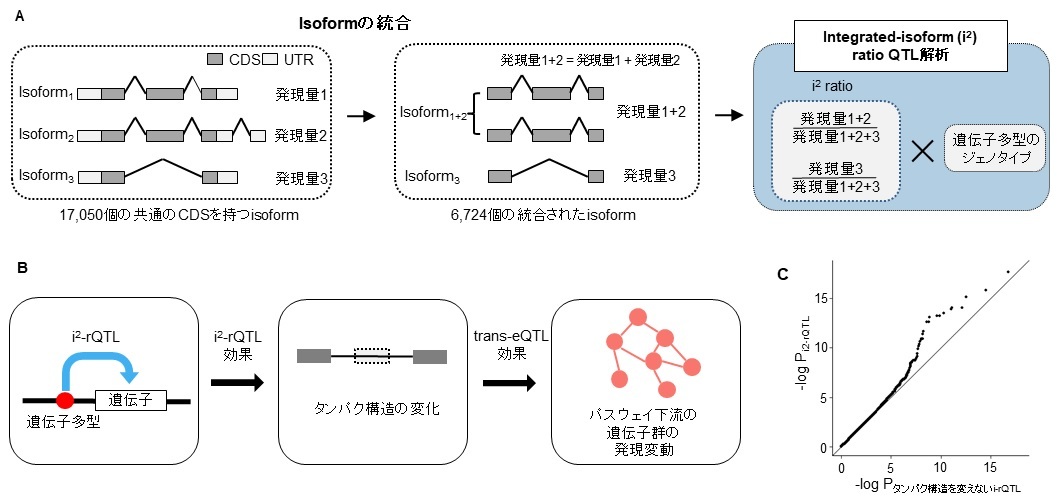
図1. i2-rQTL (integrated-isoform ratio QTL)解析
もう一つの戦略として、研究グループは、CDS incomplete isoform (CDSI)に注目したスプライシングQTL解析を提案しました。CDSIとは、既知のCDSと異なる配列が断片的ながらデータベースに登録されているスプライシングアイソフォームのことです。これまでのほとんどの研究では注目されずに解析から除外されていましたが、これらの中に生物学的に意味のあるアイソフォームが含まれる可能性を考えました。そこで、第4世代シークエンシング技術を利用した、ロングリードキャプチャーRNAシーケンシングという手法を用いて、疾患と関連すると考えられるCDSIの全長を網羅的に同定しました(図2A)。全長が明らかになったスプライシングアイソフォームに対して、スプライシングQTL解析を行ったところ、29のスプライシングアイソフォームが、何らかの疾患の原因となっていることが明らかになりました。また、これらの全長が明らかになったもののアミノ酸配列からタンパク質高次構造を予測することで、その機能を推定可能であることも示しました(図2C)。
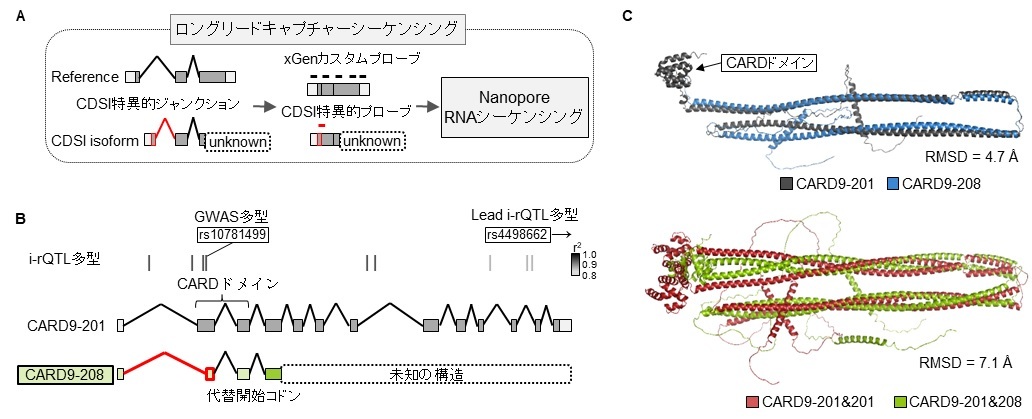
図2. CDSIに対するロングリードキャプチャーRNAシーケンシング
研究成果の意義
本研究では、複雑で解釈が難しかった無数のスプライシングイアイソフォームの中から、2つの新しいアプローチを用いて、疾患の原因となっているものに迫ることが可能であることを示しました。また今回の解析手法は、免疫疾患に限らず、全ての疾患に適応できるものです。これまで、GWASという研究手法で、延べ5万を超える遺伝子領域が疾患と関連することが明らかになってきましたが、その2~3割はスプライシングQTLが原因となっているという推計もあるため、本研究で開発した手法は様々な疾患の病態解明を加速させる可能性を秘めています。
用語解説
※1 スプライシングQTL解析・・・・・・・・一塩基多型(SNP)などの遺伝子多型が選択的スプライシングに影響を与えていることを評価する解析手法のこと。遺伝子多型のジェノタイプとスプライシングアイソフォームの発現量や発現比の相関を、統計学的に検定する。QTL(quantitative trait locus)は、遺伝学用語で、量的形質(身長、遺伝子発現量など)と関係する遺伝子座のこと。
※2スプライシングアイソフォーム・・・・・・・・遺伝子は、DNAを鋳型にRNAが転写されるが、このRNA配列はエクソン領域と、スプライシングによって切り出されるイントロン領域に分かれる。エクソン領域のみとなった成熟RNA(メッセンジャーRNA)を鋳型に、タンパク質が翻訳される。選択されるエクソン領域は、RNA分子ごとに異なるが、個々の成熟RNAをスプライシングアイソフォームと定義する。
※3ゲノムワイド関連解析(GWAS; genome-wide association study) ・・・・・・・・ゲノム上に存在する約100万個のSNPを遺伝マーカーとして用いて、疾患の原因となっている遺伝子領域を探索する手法。
※2スプライシングアイソフォーム・・・・・・・・遺伝子は、DNAを鋳型にRNAが転写されるが、このRNA配列はエクソン領域と、スプライシングによって切り出されるイントロン領域に分かれる。エクソン領域のみとなった成熟RNA(メッセンジャーRNA)を鋳型に、タンパク質が翻訳される。選択されるエクソン領域は、RNA分子ごとに異なるが、個々の成熟RNAをスプライシングアイソフォームと定義する。
※3ゲノムワイド関連解析(GWAS; genome-wide association study) ・・・・・・・・ゲノム上に存在する約100万個のSNPを遺伝マーカーとして用いて、疾患の原因となっている遺伝子領域を探索する手法。
論文情報
掲載誌:Nature Communications
論文タイトル: Splicing QTL analysis focusing on coding sequences reveals the causal mechanisms for disease susceptibility loci
DOI:https://doi.org/10.1038/s41467-022-32358-1
研究者プロフィール

山口 健介 (ヤマグチ ケンスケ) Yamaguchi Kensuke
東京医科歯科大学 生体材料工学研究所 医歯理工融合イノベーションセンター
特任助教
・研究領域
ゲノム医学、膠原病内科学
東京医科歯科大学 生体材料工学研究所 医歯理工融合イノベーションセンター
特任助教
・研究領域
ゲノム医学、膠原病内科学

高地 雄太 (コウチ ユウタ) Kochi Yuta
東京医科歯科大学 難治疾患研究所
ゲノム機能多様性分野 教授
・研究領域
ゲノム医学、膠原病内科学
東京医科歯科大学 難治疾患研究所
ゲノム機能多様性分野 教授
・研究領域
ゲノム医学、膠原病内科学
問い合わせ先
東京医科歯科大学難治疾患研究所
ゲノム機能多様性分野
〒113-8510 東京都文京区湯島1-5-45
E-mail: y-kochi.gfd[@]mri.tmd.ac.jp
<報道に関すること>
東京医科歯科大学 総務部総務秘書課広報係
〒113-8510 東京都文京区湯島1-5-45
E-mail:kouhou.adm[@]tmd.ac.jp
※E-mailは上記アドレス[@]の部分を@に変えてください。

