RBFOXとSUP-12によるRNAの協働的認識の構造基盤
Nature Structural & Molecular Biology (2014)
RBFOXファミリー制御因子と筋特異的制御因子SUP-12による線虫の線維芽細胞成長因子受容体遺伝子の筋特異的選択的スプライシング制御機構の分子基盤について紹介します.
RBFOXファミリー制御因子と筋特異的制御因子SUP-12による線虫の線維芽細胞成長因子受容体遺伝子の筋特異的選択的スプライシング制御機構の分子基盤について紹介します.
この研究成果は、本学からプレスリリースされ、サイエンスポータルで紹介されました.
この研究成果は、ライフサイエンス 新着論文レビューでも紹介しています.
この研究成果は、武蔵野大学・理化学研究所の武藤裕教授、京都大学萩原正敏教授らとの共同研究によるものです.
この研究成果は、ライフサイエンス 新着論文レビューでも紹介しています.
この研究成果は、武蔵野大学・理化学研究所の武藤裕教授、京都大学萩原正敏教授らとの共同研究によるものです.
線虫線維芽細胞成長因子(FGF)受容体遺伝子egl-15の選択的スプライシング制御
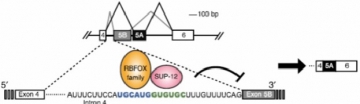
RBFOXファミリーとSUP-12によるegl-15遺伝子エクソン5Bの筋特異的な協働的抑制(Nat Struct Mol Biol, 2014より)
線虫のegl-15遺伝子は線虫で唯一のFGF受容体をコードする遺伝子で、相互排他的なエクソン5Aとエクソン5Bの使い分けによって、線虫に2種類存在するFGF(EGL-17およびLET-756)とのリガンド結合特異性が規定されます[1].
当研究室では、蛍光タンパク質GFPとRFPを用いて、egl-15遺伝子のエクソン5Aとエクソン5Bの選択をモニターできるegl-15スプライシングレポーターを作製しました[2].このレポーターでは、RFPの発現はエクソン5Aの選択を、GFPの発現はエクソン5Bの選択を反映します[2].
このレポーター線虫を用いてレポーターの色が異常になる変異体のスクリーニングを行い、筋組織特異的選択的スプライシングの制御因子として、RBFOXファミリーRNA結合タンパク質ASD-1 (Alternative-Splicing-Defective-1)およびFOX-1と筋特異的RNA結合タンパク質SUP-12を同定していました(右図)[2,3].
当研究室では、蛍光タンパク質GFPとRFPを用いて、egl-15遺伝子のエクソン5Aとエクソン5Bの選択をモニターできるegl-15スプライシングレポーターを作製しました[2].このレポーターでは、RFPの発現はエクソン5Aの選択を、GFPの発現はエクソン5Bの選択を反映します[2].
このレポーター線虫を用いてレポーターの色が異常になる変異体のスクリーニングを行い、筋組織特異的選択的スプライシングの制御因子として、RBFOXファミリーRNA結合タンパク質ASD-1 (Alternative-Splicing-Defective-1)およびFOX-1と筋特異的RNA結合タンパク質SUP-12を同定していました(右図)[2,3].
ASD-1とSUP-12によるegl-15 mRNA前駆体の協働的認識の構造基盤
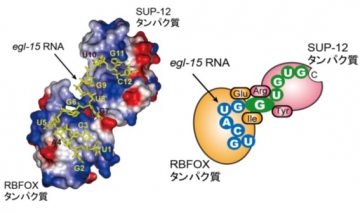
ASD-1とSUP-12により協働認識されたegl-15遺伝子のRNAの立体構造の表面電荷モデル(左)と模式図(右)(Nat Struct Mol Biol, 2014より)
7番目の塩基Gが2つのタンパク質にサンドイッチされている
本研究では、RBFOXファミリーとSUP-12がegl-15遺伝子のmRNA前駆体のUGCAUGGUGUGC配列を協働して特異的に認識するしくみの解明を行いました。そのために、SUP-12とRNAの2者複合体、ASD-1とSUP-12とRNAの3者複合体の構造を核磁気共鳴(NMR)法により解析し、比較しました.
その結果、SUP-12単独ではUGを含むさまざまな配列に弱く結合すること、ASD-1とSUP-12を組み合わせることでASD-1が前半のUGCAUG配列にSUP-12が後半のGUGUG配列にそれぞれ特異的に結合すること、SUP-12がGUGUG配列を認識する様式がASD-1の有無で変化し、3者複合体では2つのRNA結合タンパク質が7番目の塩基グアニン(G)をサンドイッチのように挟み込むことで2つのタンパク質の位置が固定されて複合体が安定化することが明らかとなりました(右図).
その結果、SUP-12単独ではUGを含むさまざまな配列に弱く結合すること、ASD-1とSUP-12を組み合わせることでASD-1が前半のUGCAUG配列にSUP-12が後半のGUGUG配列にそれぞれ特異的に結合すること、SUP-12がGUGUG配列を認識する様式がASD-1の有無で変化し、3者複合体では2つのRNA結合タンパク質が7番目の塩基グアニン(G)をサンドイッチのように挟み込むことで2つのタンパク質の位置が固定されて複合体が安定化することが明らかとなりました(右図).
生体におけるサンドイッチ認識の重要性
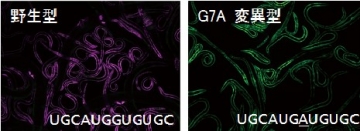
egl-15蛍光レポーター線虫(Nat Struct Mol Biol, 2014より)
赤色蛍光タンパク質(RFP)=筋肉型
緑色蛍光タンパク質(GFP)=非筋肉型
立体構造からは、RBFOXファミリーとSUP-12によるサンドイッチ認識のためには7番目の塩基がGである必要が示唆されました.
そこで、egl-15選択的スプライシングレポーターを利用して、塩基配列の改変実験を行い、標的RNAのサンドイッチ認識のためには7番目の塩基がGでなければならないこと、UGCAUG配列とGUGUG配列が隣り合っていなければならないことを確認しました(右図).
そこで、egl-15選択的スプライシングレポーターを利用して、塩基配列の改変実験を行い、標的RNAのサンドイッチ認識のためには7番目の塩基がGでなければならないこと、UGCAUG配列とGUGUG配列が隣り合っていなければならないことを確認しました(右図).
UGCAUGGUGUG配列によるスプライシング制御
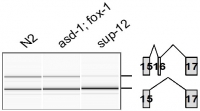
cle-1遺伝子エクソン16のスプライシングパターンの解析(Nat Struct Mol Biol, 2014より)
野生型(N2)、asd-1;fox-1二重変異体、sup-12変異体.
上記のegl-15選択的スプライシングレポーターを利用した解析で、RBFOXファミリーとSUP-12が協働して認識する配列はUGCAUGG(U/C)GUGであることがわかりました.そこで、この配列を手がかりに、他にRBFOXファミリーとSUP-12によって協働的に制御されているmRNA前駆体を探索しました.
C. elegansのゲノム上には、TGCATGG(T/C)GTG配列が47箇所あり、そのうち14箇所がタンパク質をコードする遺伝子のセンス鎖にありました.そのうちの1つ、コラーゲンXV型およびXVIII型をコードするcle-1遺伝子のイントロン16にUGCAUGGUGUG配列が線虫でたいへんよく保存されており、RT-PCRで解析したところ、エクソン16のスプライシングがRBFOXファミリーとSUP-12によって促進されることを確認しました(右図).
これは、ゲノムの塩基配列情報のみを基にRBFOXファミリーとSUP-12によって共通して制御され選択的スプライシングの標的エクソンを同定した初めての例となり、RNAの協働的認識機構を明らかにしたことで選択的スプライシングパターンをより正確に予測できるようになったことを実証するものとなりました.
C. elegansのゲノム上には、TGCATGG(T/C)GTG配列が47箇所あり、そのうち14箇所がタンパク質をコードする遺伝子のセンス鎖にありました.そのうちの1つ、コラーゲンXV型およびXVIII型をコードするcle-1遺伝子のイントロン16にUGCAUGGUGUG配列が線虫でたいへんよく保存されており、RT-PCRで解析したところ、エクソン16のスプライシングがRBFOXファミリーとSUP-12によって促進されることを確認しました(右図).
これは、ゲノムの塩基配列情報のみを基にRBFOXファミリーとSUP-12によって共通して制御され選択的スプライシングの標的エクソンを同定した初めての例となり、RNAの協働的認識機構を明らかにしたことで選択的スプライシングパターンをより正確に予測できるようになったことを実証するものとなりました.
本研究成果の意義
RNAのプロセシングの制御は、RNAの塩基配列を特異的に認識して結合するさまざまなRNA結合タンパク質が担っています.ところが、個々のRNA結合ドメインが認識して結合するのは4~6塩基程度の短くてあいまいな配列であることが知られており、このようなRNA結合タンパク質でたくさんの種類のRNAの加工を正確に制御できるしくみはよく解っていませんでした.
本研究は、2つのRNA結合タンパク質が協働することで、個々のタンパク質が単独で認識する様式の場合よりも長い配列をより正確に認識するしくみがあることを初めて明らかにしました.
たくさんの遺伝子のRNAの加工が組織や細胞の種類に応じて巧妙に制御されるしくみは未だに完全には解明されておらず、RNA加工の「細胞暗号」と呼ばれています.これまでは、RNA結合タンパク質は個別に研究されてきました.しかし、本研究成果は、RNA結合タンパク質のさまざまな組み合わせによりRNAが協働的に認識されるしくみを解明していくことが「細胞暗号」の完全な解読のためには必須であることを示しています.
本研究で行われたように、遺伝学的解析と構造学的解析さらに生物情報学的解析を組み合わせることで、生体におけるRNA加工の「細胞暗号」の体系的な解読につながっていくことが期待されます.
本研究は、2つのRNA結合タンパク質が協働することで、個々のタンパク質が単独で認識する様式の場合よりも長い配列をより正確に認識するしくみがあることを初めて明らかにしました.
たくさんの遺伝子のRNAの加工が組織や細胞の種類に応じて巧妙に制御されるしくみは未だに完全には解明されておらず、RNA加工の「細胞暗号」と呼ばれています.これまでは、RNA結合タンパク質は個別に研究されてきました.しかし、本研究成果は、RNA結合タンパク質のさまざまな組み合わせによりRNAが協働的に認識されるしくみを解明していくことが「細胞暗号」の完全な解読のためには必須であることを示しています.
本研究で行われたように、遺伝学的解析と構造学的解析さらに生物情報学的解析を組み合わせることで、生体におけるRNA加工の「細胞暗号」の体系的な解読につながっていくことが期待されます.
文献
1. Goodman SJ, Branda CS, Robinson MK, Burdine RD, Stern MJ. (2003) Alternative splicing affecting a novel domain in the C. elegans EGL-15 FGF receptor confers functional specificity. Development 130, 3757-3766.
2. Kuroyanagi H, Kobayashi T, Mitani S, Hagiwara M. (2006) Transgenic alternative-splicing reporters reveal tissue-specific expression profiles and regulation mechanisms in vivo. Nat Methods 3, 909-915.
3. Kuroyanagi H, Ohno G, Mitani S, Hagiwara M. (2007) The Fox-1 family and SUP-12 coordinately regulate tissue-specific alternative splicing in vivo. Molecular and Cellular Biology 27, 8612-8621.
2. Kuroyanagi H, Kobayashi T, Mitani S, Hagiwara M. (2006) Transgenic alternative-splicing reporters reveal tissue-specific expression profiles and regulation mechanisms in vivo. Nat Methods 3, 909-915.
3. Kuroyanagi H, Ohno G, Mitani S, Hagiwara M. (2007) The Fox-1 family and SUP-12 coordinately regulate tissue-specific alternative splicing in vivo. Molecular and Cellular Biology 27, 8612-8621.

