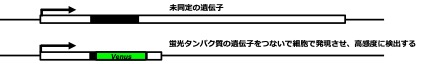
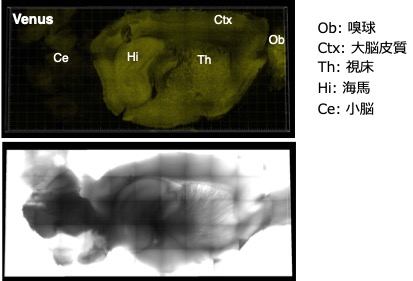
ヒトゲノムに含まれる遺伝子数は約2万と言われていますが、これはゲノム全体の1.5%を占めるに過ぎず、残る部分に未同定の遺伝子が多数残されている可能性があります。ネオゲノム研究開発ユニットは、タンパク質をコードし遺伝子機能を持つ可能性のあるDNA配列を同定し、その機能を明らかにすることを目指しています。
未来医療コンソーシアムでは個人のゲノム情報に基づいた疾患の診断、治療などを行うことが到達目標の1つとなっていますが、そのためには疾患に関係する遺伝子の同定が最も重要な課題です。本ユニットの役割は、この戦略を有効に活かすためにヒトゲノムにおける正確な遺伝子数を推定し、未同定の疾患関連遺伝子を把握することです。
今後のゲノム医療においてAIは非常に重要な役割を果たすことになります。研究者が行うべきことは、完全な情報をAIが解析できる状況を作ることです。ヒトゲノム機能には未知の部分が多く残されており、これをどれだけ完全に理解できるかは、まさに科学者の力量にかかっている部分であると考えています。
大学のプロジェクトとして、AIを活用したゲノム機能解析は10年後、20年後を見据えた戦略の一つであり、未来医療のカギを握る部分であると考えています。また、ヒトを生物学的に理解するという、生物学の究極の目標に近づくための研究であり、若い研究者たちにも是非、参入してほしいと考えています。