1.転写の過程
2.リボソームとtRNA
3.翻訳の過程
4.翻訳されたタンパク質の行方
関連するサイトとリンク(このページへ戻るときはブラウザーの戻るを選んでください) 更新日:2003/05/21
第8章ではDNAからタンパク質までの大まかな道筋を描いたが、実際にタンパク質が細胞の中でどのように合成されるかについては深く立ち入らなかった。この章ではmRNAへの転写からタンパク質合成までの過程をもう少し詳しく見ていこう。
細胞内でのタンパク質合成の過程は、核の中でおこるDNAからmRNAへの転写と、核外へ出たmRNAを使っておこなわれる翻訳の過程に分けられる。
http://chemed.chem.purdue.edu/genchem/topicreview/bp/1biochem/synthesis9.html
DNAに遺伝情報が塩基の4文字で書かれている事は分かったが、この情報がタンパク質の合成に利用されるためにはmRNAへ転写されなければならない。一つの遺伝子は一本のポリペプチド鎖をコードしているのだから始まりと終わりがあり、これに対応する開始コドンと終止コドンがある。つまり、DNAには開始の花文字から句点までの一まとまりのセンテンスが、カセットテープに複数の曲が録音されているように、線状に並んでいることになる。それでは必要な遺伝子の情報の読み出し、すなわち頭出しをどのようにしておこなっているのだろうか。
1)RNA
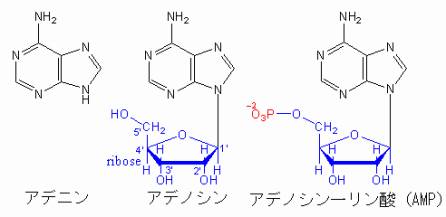
ここでRNAのことに少し触れなくてはならない。RNAはDNAと同じく核酸の一種で次の3点がDNAと異なる点である。
1)DNAでは五炭糖がデオキシリボースだが、RNAではリボースである。
2)塩基の4種類がTACGではなく、UACGである。
3)二重ラセン構造を取らず、一本鎖のままである。
リボースとデオキシリボースの違いは、2’に水酸基がついているか、いないかである。また、チミン(T)とウラシル(U)の違いは、メチル基のあるなしで、UでもTと同じように、Aとの間に相補的な水素結合が2本できる。

2)RNAポリメラーゼ
DNAからmRNAへの転写は、酵素であるRNAポリメラーゼによって触媒される。RNAポリメラーゼは、DNAの二重ラセンをほどきながら、二本鎖のうち鋳型となる鎖の塩基の配列を読んで、これと相補的な塩基をもったヌクレオチドを次々と呼び込んで結合をつくっていく。RNAの鎖の伸長は必ず5’→3’の方向におきるので、鋳型鎖の配列にしたがってこの方向に塩基をつないでいくと、コード鎖と同じ塩基の配列(ただしTはUとなる)をもったmRNAができることになる。
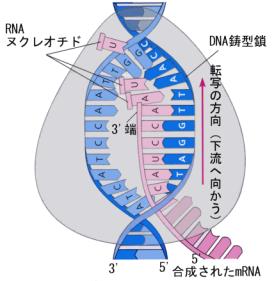
DNA (5'
→ 3')
ATGGAATTCTCGCTC (コード鎖、sense
strand)
二本鎖 (3' ← 5') TACCTTAAGAGCGAG (鋳型鎖、antisense strand)
↓(転写)
転写された (5'
→ 3')
AUGGAAUUCUCGCUC
一本鎖のmRNA
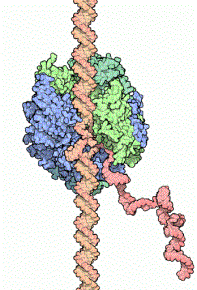
RNAポリメラーゼは複雑な構造をした酵素タンパク質で、12個のポリペプチド鎖から構成されている。上左の図のように二本鎖のDNAを包み込むように結合し、DNAに沿って動きながら右下に見えるピンク色のmRNAを合成していく。
3)プロモーターと転写の開始点
それではどうやって、頭出しをしているのだろうか。その秘密は、DNAの塩基配列にある。DNAの塩基配列にはアミノ酸配列をコードしている領域と、転写の調節に関与する領域がある。調節領域には、タンパク質が結合するための目印が塩基の4文字で書きこまれている。
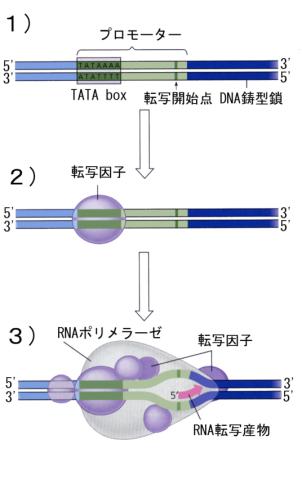 RNAポリメラーゼが結合するこの領域は、開始コドン(ATG)のすぐ上流にあり、プロモーターと呼ばれている。真核生物では、このプロモーター領域のうち、開始コドン上流30塩基を中心にTATAAAという配列が共通して存在する。そのため、この領域のことをTATA
boxとかホグネス配列とか読んでいる(右図の1))。
RNAポリメラーゼが結合するこの領域は、開始コドン(ATG)のすぐ上流にあり、プロモーターと呼ばれている。真核生物では、このプロモーター領域のうち、開始コドン上流30塩基を中心にTATAAAという配列が共通して存在する。そのため、この領域のことをTATA
boxとかホグネス配列とか読んでいる(右図の1))。
転写の開始は、まずTATA boxに転写因子(タンパク質)が結合することから始まる(右図の2))。これを目印に次にRNAポリメラーゼ(やその他の転写因子)が結合し、転写開始点からの転写が開始される(右図の3))。
RNAポリメラーゼが、転写終了を示すターミネーターまで達すると、転写は終了する。
プロモーターはRNAポリメラーゼの着地点であるとともに、この酵素がDNA上を滑っていく方向も規定する。したがって、二本鎖のうちのどちらが鋳型鎖になるかは、プロモーターの配置によって決まる。
4)プロセシング
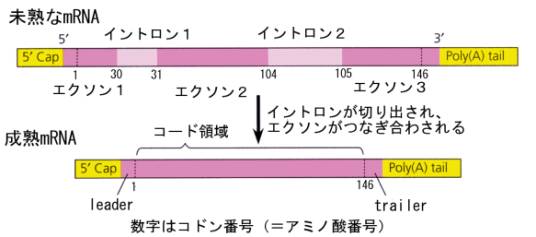
真核生物の場合は、転写されたままではmRNAとしては未完成で、タンパク質合成に使うことはできない。転写産物が成熟したmRNAになるために、いろいろな修飾を受ける。この修飾の過程をプロセシングと呼んでいる。
プロセシングの一つは余分な構造の付加で、5’側にCAP構造、3’側にAが連続したpolyA tailが付加される。したがって、開始コドンから終了コドンまでのコード領域は、この間に含まれていることになる。
もう一つはスプライシングと呼ぶ過程である。これは、真核生物の遺伝子では、開始コドンから終了コドンまでの間に、タンパク質のアミノ酸配列の情報をもった領域と情報をもたない領域が混在しているからである。前の領域をエクソンと呼び、後者をイントロンと呼んでいる。RNAポリメラーゼは、転写開始点からターミネーターまで、連続して転写してしまうので、エクソンもイントロンも両方とも含んだ転写産物ができてしまう。
そのため、アミノ酸配列の情報をもたないイントロン部分を切り出す必要がある。この切り出しの過程がスプライシングである。テレビ番組を録画したあと、広告の部分を編集によって切り取って、本編だけをつなぎ合わせるのと同じことである。
これらの過程は核の中でおこる。こうして、最終的に成熟したmRNAが核膜孔を通って、サイトゾールに送られる。
次の塩基の配列はヒトβグロビンの遺伝子である。赤色の3箇所を含む最初の部分がプロモーター領域で、3番目の赤い部分がTATA box、水色のACATTが転写開始点である。紺色で示したATGは開始コドンで、マゼンダ色の部分がエクソンで3つあり、間に挟まれた2箇所がイントロンである。3番目のエクソンの紺色のTAAが終止コドンである。その後の水色の部分は転写されるが翻訳されない部分で、赤色の部分にpolyA tailが付加される。
CCCTGTGGAGCCACACCCTAGGGTTGGCCAATCTACTCCCAGGAGCAGGGA
GGGCAGGAGCCAGGGCTGGGCATAAAAGTCAGGGCAGAGCCATCTATTGCT
TACATTTGCTTCTGACACAACTGTGTTCACTAGCAACCTCAAACAGACACC
ATGGTGCACCTGACTCCTGAGGAGAAGTCTGCCGTTACTGCCCTGTGGGGC エクソン1(コドン1−30)
AAGGTGAACGTGGATGAAGTTGGTGGTGAGGCCCTGGGCAGGTTGGTATCA イントロン1
AGGTTACAAGACAGGTTTAAGGAGACCAATAGAAACTGGGCATGTGGAGAC
AGAGAAGACTCTTGGGTTTCTGATAGGCACTGACTCTCTCTGCCTATTGGT
CTATTTTCCCACCCTTAGGCTGCTGGTGGTCTACCCTTGGACCCAGAGGTT エクソン2(コドン31−104)
CTTTGAGTCCTTTGGGGATCTGTCCACTCCTGATGCTGTTATGGGCAACCC
TAAGGTGAAGGCTCATGGCAAGAAAGTGCTCGGTGCCTTTAGTGATGGCCT
GGCTCACCTGGACAACCTCAAGGGCACCTTTGCCACACTGAGTGAGCTGCA
CTGTGACAAGCTGCACGTGGATCCTGAGAACTTCAGGGTGAGTCTATGGGA イントロン2
CCCTTGATGTTTTCTTTCCCCTTCTTTTCTATGGTTAAGTTCATGTCATAG
GAAGGGGAGAAGTAACAGGGTACAGTTTAGAATGGGAAACAGACGAATGAT
TGCATCAGTGTGGAAGTCTCAGGATCGTTTTAGTTTCTTTTATTTGCTGTT
CATAACAATTGTTTTCTTTTGTTTAATTCTTGCTTTCTTTTTTTTTCTTCT
CCGCAATTTTTACTATTATACTTAATGCCTTAACATTGTGTATAACAAAAG
GAAATATCTCTGAGATACATTAAGTAACTTAAAAAAAAACTTTACACAGTC
TGCCTAGTACATTACTATTTGGAATATATGTGTGCTTATTTGCATATTCAT
AATCTCCCTACTTTATTTTCTTTTATTTTTAATTGATACATAATCATTATA
CATATTTATGGGTTAAAGTGTAATGTTTTAATATGTGTACACATATTGACC
AAATCAGGGTAATTTTGCATTTGTAATTTTAAAAAATGCTTTCTTCTTTTA
ATATACTTTTTTGTTTATCTTATTTCTAATACTTTCCCTAATCTCTTTCTT
TCAGGGCAATAATGATACAATGTATCATGCCTCTTTGCACCATTCTAAAGA
ATAACAGTGATAATTTCTGGGTTAAGGCAATAGCAATATTTCTGCATATAA
ATATTTCTGCATATAAATTGTAACTGATGTAAGAGGTTTCATATTGCTAAT
AGCAGCTACAATCCAGCTACCATTCTGCTTTTATTTTATGGTTGGGATAAG
GCTGGATTATTCTGAGTCCAAGCTAGGCCCTTTTGCTAATCATGTTCATAC
CTCTTATCTTCCTCCCACAGCTCCTGGGCAACGTGCTGGTCTGTGTGCTGG エクソン3(コドン105−146)
CCCATCACTTTGGCAAAGAATTCACCCCACCAGTGCAGGCTGCCTATCAGA
AAGTGGTGGCTGGTGTGGCTAATGCCCTGGCCCACAAGTATCACTAAGCTC
GCTTTCTTGCTGTCCAATTTCTATTAAAGGTTCCTTTGTTCCCTAAGTCCA
ACTACTAAACTGGGGGATATTATGAAGGGCCTTGAGCATCTGGATTCTGCC
TAATAAAAAACATTTATTTTCATTGCAATGATGTATTTAAATTATTTCTGA
ATATTTTACTAAAAAGGGAATGTGGGAGGTCAGTGCATTTAAAACATAAAG
AAATGAAGAGCTAGTTCAAACCTTGGGAAAATACACTATATCTTAAACTCC
ATGAAAGAAGGTGAGGCTGCAAACAGCTAATGCACATTGGCAACAGCCCTG
ATGCCTATGCCTTATTCATCCCTCAGAAAAGGATTCAAGTAGAGGCTTGAT
TTGGAGGTTAAAGTTTTGCTATGCTGTATTTTACATTACTTATTGTTTTAG
CTGTCCTCATGAATGTCTTTTCACTACCCATTTGCTTATCCTGCATCTCTC
AGCCTTGACTCCACTCAGTTCTCTTGCTTAGAGATACCACCTTTCCCCTGA
AGTGTTCCTTCCATGTTTTACGGCGAGATGGTTTCTCCTCGCCTGGCCACT
CAGCCTTAGTTGTCTCTGTTGTCTTATAGAGGTCTACTTGAAGAAGGAAAA
ACAGGG GGCATGGTTTGACT……
遺伝情報がDNAに書き込まれているからといって、すべての情報が転写されて、タンパク質になるわけではない。もしもそうだったら、すべての細胞が同じ形をして、同じ機能を発揮してしまうことになる。そうならないのは、転写の調節がおこなわれているからである。また転写される場合でも、転写の量的な調節がおこることが多い。そのため、同じ遺伝子組成の生物でも、置かれた環境によって表現形に量的な変異があらわれることがある。このような変異(環境変異とか彷徨変異と呼ばれる)は遺伝子突然変異とは異なり、遺伝はしない。
1)リボソーム
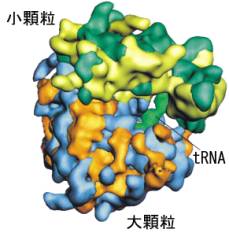 すでに第7章で述べたように、タンパク質の合成の場はリボソームである。リボソームはrRNAとたんぱく質の複合体で、大顆粒と小顆粒からなるダルマ型をしているが、それぞれの顆粒の表面や顆粒の連結部が複雑なかたちをしていて、そこにmRNAやtRNAが結合できる場所になっている。これらの結合部位のおかげで、mRNAの塩基の配列情報とアミノ酸の配列情報の対応を、アダプターであるtRNAのはたらきを借りて取れるようになっている。簡単に言えばリボソームは、塩基語からアミノ酸語への翻訳機なのである。
すでに第7章で述べたように、タンパク質の合成の場はリボソームである。リボソームはrRNAとたんぱく質の複合体で、大顆粒と小顆粒からなるダルマ型をしているが、それぞれの顆粒の表面や顆粒の連結部が複雑なかたちをしていて、そこにmRNAやtRNAが結合できる場所になっている。これらの結合部位のおかげで、mRNAの塩基の配列情報とアミノ酸の配列情報の対応を、アダプターであるtRNAのはたらきを借りて取れるようになっている。簡単に言えばリボソームは、塩基語からアミノ酸語への翻訳機なのである。
左の図で小顆粒の緑と黄緑色の部分はそれぞれタンパク質とrRNA、大顆粒の黄色と水色はタンパク質とrRNAで、このように入り組んだ複合体となっている。
さらに詳しくは下記サイトを参照してください。
http://ntri.tamuk.edu/cell/ribosomes.html
http://www.blc.arizona.edu/marty/411/Modules/ribtRNA.html
2)tRNAの構造
クリックのアダプター仮説は実に秀逸な仮説だった。アダプターという考えによって塩基の配列とアミノ酸の配列の対応が取れることになるからである。アダプターの実体を探したところそれがtRNAであることが明らかになった。
アダプターであるtRNAには2つの機能が必要である。1つはmRNAのコドンを認識すること、もう1つはアミノ酸を結合することである。しかもその両者は正しく対応しなければならない。
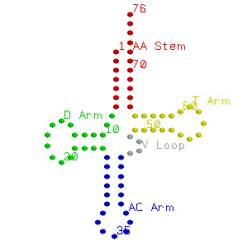 tRNAは、73から93個のヌクレオチド(対応するアミノ酸によって異なる)が連結した構造をしているが、一本鎖の中に相補的な塩基間の水素結合が4ヶ所かあるために、その部分が柄となった三つ葉のクローバ形をしている。大部分のヌクレオチドは共通しているが、下の図でACアームと書いてある部分にアンチコドンと呼ばれる連続した3つのヌクレオチドが含まれていて、この部分がmRNAを認識するところである。3’側に共通のCCAという配列があり、その先にアンチコドンに対応した特定のアミノ酸が結合している。
tRNAは、73から93個のヌクレオチド(対応するアミノ酸によって異なる)が連結した構造をしているが、一本鎖の中に相補的な塩基間の水素結合が4ヶ所かあるために、その部分が柄となった三つ葉のクローバ形をしている。大部分のヌクレオチドは共通しているが、下の図でACアームと書いてある部分にアンチコドンと呼ばれる連続した3つのヌクレオチドが含まれていて、この部分がmRNAを認識するところである。3’側に共通のCCAという配列があり、その先にアンチコドンに対応した特定のアミノ酸が結合している。
実際には、上の図の赤い柄の部分は右に折れ曲がっているし、他の部分もラセン構造をとっているので、全体としては下図のように、L字をひっくり返した形をしている。

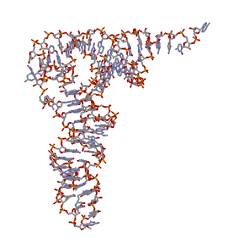
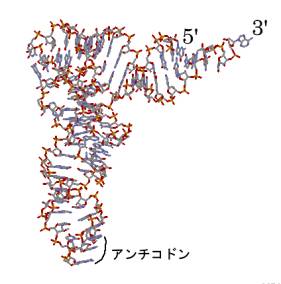
アダプターの働きをするアンチコドンとアミノ酸の結合部は、この逆L字形の両端にあり、長い棒の先にアンチコドン部があり、短い棒の先端にアミノ酸が結合する。上の図はフェニルアラニン(Phe)のtRNAである。3番目の図は5’と3’がわかりやすくなるように少し回転させてある。3’側に先端にPheが結合する。アンチコドンと書いてある部分の3段の塩基が、mRNAのコドンと相補的に結合するアンチコドンである。
20種のアミノ酸の運搬には、それぞれのアミノ酸に対応したtRNAが必要だが、コドンのそれぞれに対応してtRNAがあるわけではない。多くのアミノ酸では複数のコドンと対応していて、3番目の塩基はどの塩基でもよいようになっている。3番目の塩基に対応するアンチコドンの1番目の塩基は修飾を受け、4種の塩基に対応できるようになっている。
http://info.bio.cmu.edu/Courses/BiochemMols/tRNA_Tour/tRNAMain.htm
http://www.tulane.edu/~biochem/nolan/lectures/rna/trnaz.htm
http://anx12.bio.uci.edu/~hudel/bs99a/lecture21/
3)tRNAにアミノ酸を付加する
細胞の中では常にタンパク質合成が起こっているので、原料となるtRNAは常に供給されている必要がある。食べ物から取り込んで分解されたアミノ酸は細胞に供給される。このアミノ酸は、アミノアシルtRNA合成酵素という酵素によってtRNAに結合され、合成の場に次々と供給される。アミノアシルtRNA合成酵素は、逆L字形のtRNAとピッタリと結合してアンチコドン部を読み取り、これに合うアミノ酸を取り込んで3’側のCAAのAにリン酸を介して結合をつくる。
http://www.rpi.edu/dept/bcbp/molbiochem/MBWeb/mb2/part1/trna.htm
http://www.rpi.edu/dept/bcbp/molbiochem/MBWeb/mb2/part1/chime/aatrna/trna-index.html(アミノアシルtRNA合成酵素)
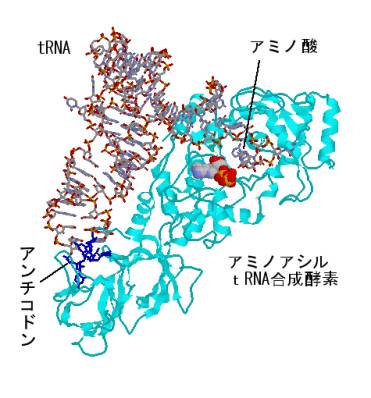
さあ、これでmRNAの情報と、tRNAの原料と、の合成の場であるリボソームが用意された。あとはどのように合成が進んでいくかという点だけである。翻訳の過程は、ステップバイステップでひとつづつアミノ酸をペプチド結合でつないでいく過程である。それがリボソームの表面で起こるのである。
 1)リボソームの座席表
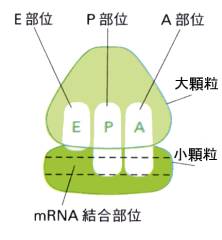
1)リボソームの座席表
リボソームのコンピュータモデルを上の図で示したが、話を簡単にするために下のようにさらに模式的に描くことにする。話の都合上、この図ではダルマさんをひっくり返して、小顆粒を下に描いている。
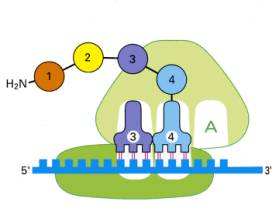
小顆粒にはmRNAの結合部位があり、小顆粒と大顆粒にまたがって、3つの凹みがある。スポーツタイプの自動車の座席によくある、バケットシートを想像するとよい。右から順にA部位、P部位、E部位と並んでいる。A部位のAはアミノアシルtRNAの略であり、アミノ酸を3’末端に結合したtRNAだけが座ることができるシートである。次のP部位のPはペプチジルtRNAのPであり、ペプチド鎖を結合したtRNAだけが座ることができる。さいごのEはexit出口の略で、アミノ酸もペプチド鎖も結合していないtRNAだけがリボソームから出て行くために一時的に座るシートである。
2)翻訳の開始
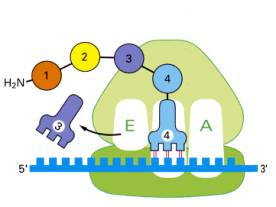
核からやってきたmRNAはまず小顆粒のmRNA結合部位の結合する。次に読み始めのコドンに対応するメチオニン(読みはじめなので少しお化粧をしている)を結合したtRNAが、アンチコドンでmRNA上の対応するコドンと結合し、ここに大顆粒が加わり、ダルマ型のリボソームとmRNAとお化粧メチオニンtRNAの複合体ができる。お化粧をしているので、このtRNAはA部位ではなくP部位に座る。
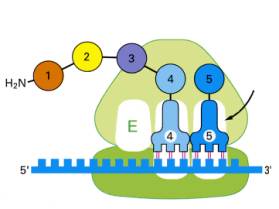
右隣のA部位は空席となっているので、ここにメチオニンの次のコドンに対応するアンチコドンをもったtRNAが結合する。メチオニンはtRNAから離れて、隣のA部位のtRNAに結合しているアミノ酸とペプチド結合を作る。そうするとP部位に座っていたtRNAはもはや何も結合していないので、ルールに従いE部位に移りリボソームから出て行く。
A部位に座っていたtRNAはジペプチドを結合しているので、やはりルールに従い左隣のP部位に移る。こうして、P部位にジペプチドを結合したtRNAが結合し、A部位が空席になったリボソームとなる。
3)ペプチドの伸長
あとはこれの繰り返しが起こり、アミノ酸が次々と付加されてペプチド鎖の伸長がおこる。下の図は1がお化粧メチオニンで2がその次のアミノ酸で、以下さらに3つのアミノ酸が付加される様子を模式的に描いたものである。
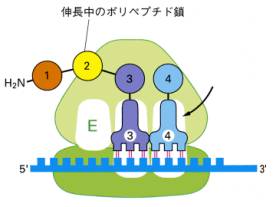
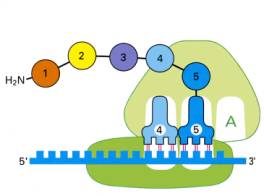
リボソームが終止コドンまでくると、終止因子と呼ぶタンパク質分子がこのコドンを認識してA部位に座る。その結果、翻訳はそれ以上進まず、完成したポリペプチド鎖が切り離されるとともに、mRNA、大小のリボソーム顆粒、RNAがバラバラになる。こうして、DNAの遺伝情報(コドン)を正確にアミノ酸に置き換えたポリペプチド鎖が完成する。読み取り開始から終わりまで、平均して20秒から60秒ほどかかる。
ふつうは、上に述べたように1本のmRNAに1個のリボソームがついて、1本のポリペプチド鎖ができるのではなく、1本のmRNAにたくさんのリボソームがついて次々とポリペプチド鎖を合成して行く。
このように1本のmRNAにたくさんのダルマさんがつながったようなものをポリリボソーム(あるいは単にポリソーム)と呼んでいる。細胞の内部で使われるタンパク質は、翻訳が終わるとタンパク質の高次構造をとって機能を持つようになり、サイトゾールへ供給される。
http://www.cell.com/content/article/abstract?uid=PIIS0092867402006190(アドバンスト)
3節で述べたタンパク質は、細胞内部で使われるハウスキーピング(house-keeping)遺伝子から翻訳されるタンパク質であったが、消化酵素やホルモンのように細胞の外部へ分泌されるタンパク質、あるいは膜タンパク質の場合は、これとは少し異なる過程をたどる。
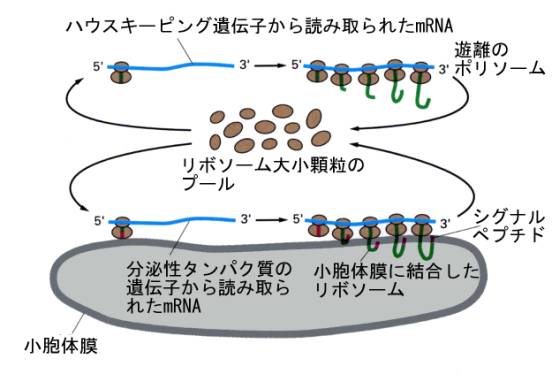
上の図にあるように、分泌性タンパク質や膜タンパク質遺伝子の最初の部分には、特別な指令が書き込まれている。この部分が翻訳されると、メチオニンから始まる20から30個のアミノ酸からなる特別なペプチド、シグナルペプチドと呼ばれる配列になる。このシグナルペプチドは、特別なやり方で小胞体の表面に開いた孔の縁に結合する。その結果、合成されたポリペプチド鎖は、孔を通って小胞体の腔所の内部へ入っていく。
小胞体内に入ると、シグナルペプチドは切り離され、糖が付加されたりして、すでに述べたようにゴルジ体へ送られる。
細胞内でつくられたタンパク質は、小胞体へ入るものの他にも、その使用目的に応じた適切な場所に送り込まれるために、ソートされる。たとえば、リボソームの構成要素となるタンパク質は核へ向かい、核膜孔を通って核内に入り、そこでrRNAと複合体をつくる。また、ミトコンドリアに入ってエネルギー産生を担う酵素となるものもある。ソーティングの詳しいことは、この学習の範囲を越えているので、ここでは省略する。
![]() この章のpdfファイルをダウンロードするには、左の「Adobe」のアイコンを右クリックして、ファイルを保存を選んで、自分のパソコンにダウンロードしてください。
この章のpdfファイルをダウンロードするには、左の「Adobe」のアイコンを右クリックして、ファイルを保存を選んで、自分のパソコンにダウンロードしてください。
![]() ブロードバンド接続の場合で、ワードのファイルを望む人は、「W」のアイコンを右クリックしてください。
ブロードバンド接続の場合で、ワードのファイルを望む人は、「W」のアイコンを右クリックしてください。