丂丂侾丏傕偆彮偟徻偟偄堚揱巕敪尰偺偼側偟
丂丂丂丂丂侾乯揮幨偲僾儘儌乕僞
丂丂丂丂丂俀乯尨妀惗暔偺応崌
丂丂丂丂丂俁乯恀妀惗暔偺応崌
丂丂俀丏堚揱巕岺妛
丂丂 侾乯僞儞僷僋幙傛傝DNA偺傎偆偑埖偄偑娙扨
俀乯珲亖惂尷峺慺偲屝亖俢俶俙儕僈乕僛
俁乯僾儔僗儈僪儀僋僞乕偲儔僀僽儔儕乕
係乯僾儘乕僽乮probe乯偺棙梡
俆乯們俢俶俙儔僀僽儔儕乕
俇乯俢俶俙墫婎攝楍偺寛掕
俈乯堚揱巕摫擖摦暔乮transgenic organism乯
丂娭楢偡傞僒僀僩偲儕儞僋乮偙偺儁乕僕傊栠傞偲偒偼僽儔僂僓乕偺栠傞傪慖傫偱偔偩偝偄乯 丂丂堚揱妛揹巕攷暔娰(崙棫堚揱妛尋媶強嶌惉乯 峏怴擔丗2002/07/04
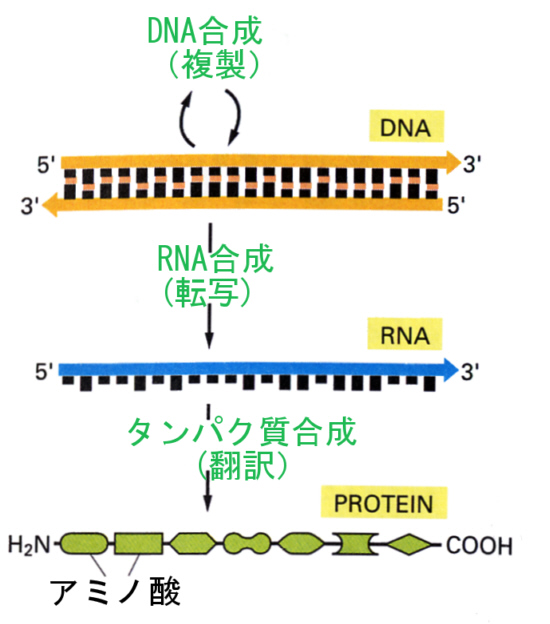
丂乽妀亅堚揱忣曬偺憅屔乿偺儁乕僕偱愼怓懱傪峔惉偡傞俢俶俙偺墫婎偺攝楍偑堚揱偺埫崋偱丄僐僪儞乮俁偮偺墫婎乯偑侾偮偺傾儈僲巁傪巜掕偟丄偟偨偑偭偰墫婎偺攝楍偵傛偭偰傾儈僲巁偺堦師峔憿乮攝楍)偑寛掕偝傟傞偙偲傪妛傫偩丅DNA偐傜僞儞僷僋幙傊偺堚揱忣曬偺棳傟偼師偺恾偱昞偡偙偲偑偱偒傞丅

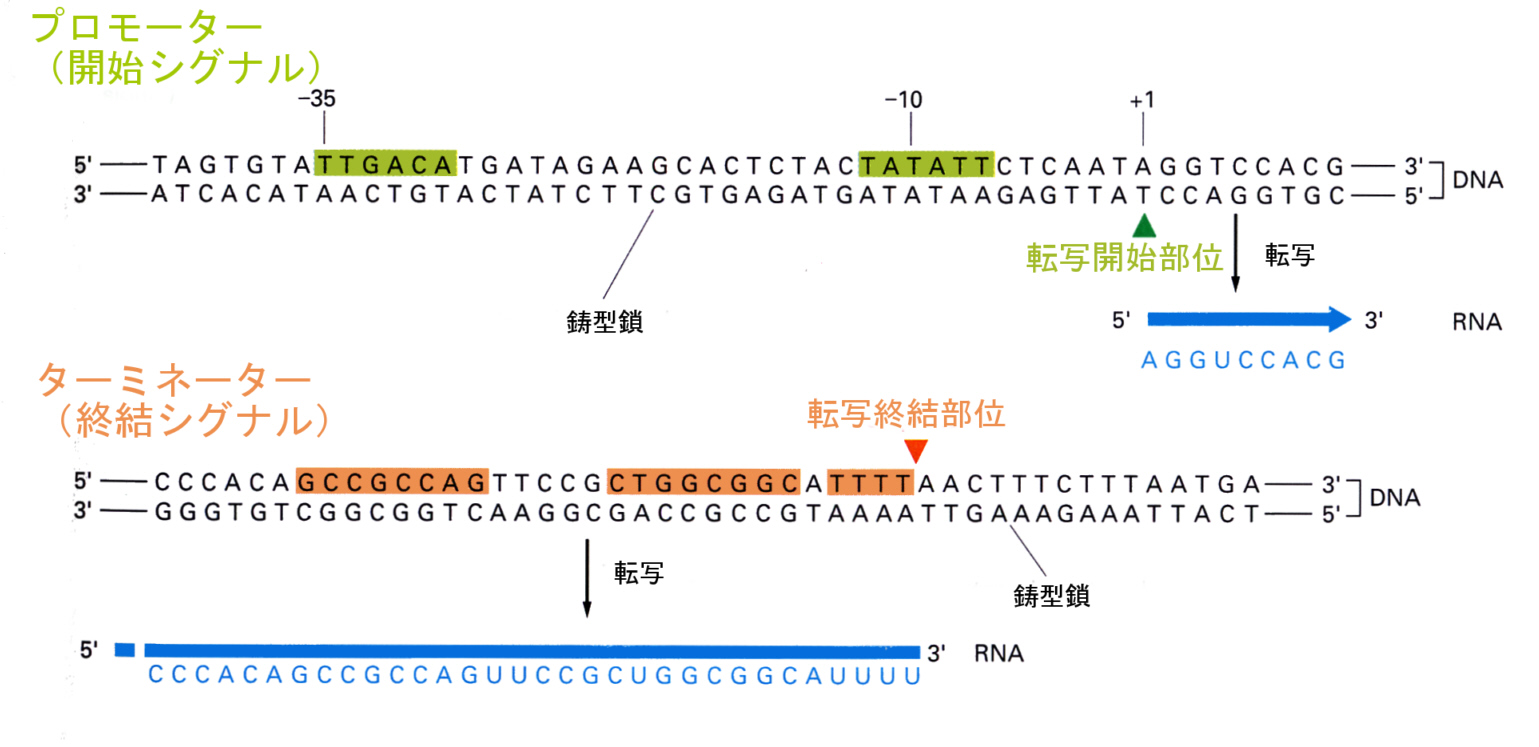
丂揮幨偵傛偭偰丄DNA偺拻宆嵔偵憡曗揑側倣RNA偑嶌傜傟傞偑丄偙偺斀墳傪怗攠偡傞峺慺偼RNA億儕儊儔乕僛偱偁傞丅
丂RNA億儕儊儔乕僛偼師偺柾幃恾偑帵偡傛偆偵丄堦曽岦傊恑傒側偑傜DNA偺擇杮嵔傪傎偳偒丄拻宆嵔偲側傞俁乫仺俆乫偺墫婎偺攝楍傪撉傒庢傝側偑傜丄偙傟偲憡曗揑側儕儃僰僋儗僆僔僪嶰儕儞巁傪1屄偢偮晅壛偟偰丄RNA嵔傪俆乫仺俁乫曽岦偵怢偽偟偰偄偔丅RNA億儕儊儔乕僛偺屻曽偱偼DNA偼嵞傃擇杮嵔偺傕偳傝丄崌惉偝傟偨倣RNA偼夝棧偟偰偄偔丅
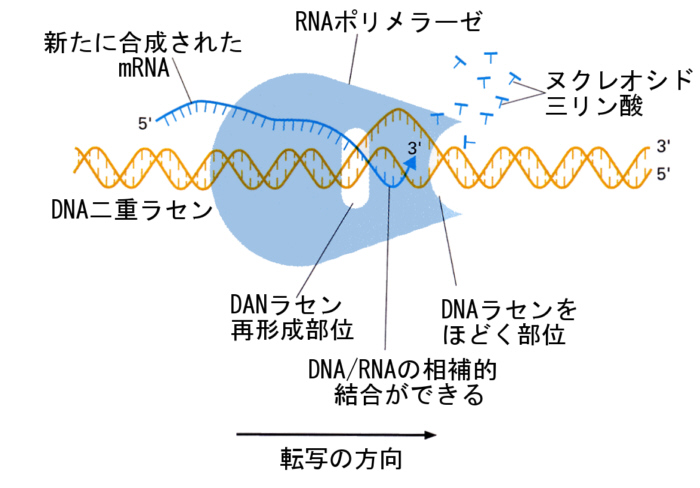
丂RNA億儕儊儔乕僛偑揮幨傪奐巒偡傞偨傔偵偼丄堚揱巕偺愭摢傪擣幆偟偰偦偙偵嫮偔寢崌偡傞昁梫偑偁傞丅偙偺揮幨奐巒偺僔僌僫儖偲側偭偰偄傞偺偑丄僾儘儌乕僞乕偲屇傇DNA偺椞堟偱偁傞丅RNA億儕儊儔乕僛偼DNA忋傪偡傋傝側偑傜堏摦偟偰偄傞偑丄僾儘儌乕僞乕椞堟偵弌夛偆偲偦偙偱嫮偔寢崌偟丄忋偵弎傋偨傛偆偵揮幨奐巒揰偐傜揮幨傪巒傔傞偙偲偵側傞丅
丂DNA忋偵偼偝傜偵丄揮幨偺廔椆傪巜帵偡傞晹埵傕懚嵼偡傞丅RNA億儕儊儔乕僛偼揮幨廔寢晹埵傑偱偔傞偲丄偦偙偱DNA傪棧傟揮幨偑廔椆偡傞丅
丂堚揱巕偼丄偙偺傛偆偵揮幨奐巒晹埵偲揮幨廔椆晹埵偵偼偝傑傟偰偄傞偺偱偁傞丅
丂偲偙傠偱丄摨偠屄懱傪峔惉偡傞嵶朎偼摨偠堚揱巕傪彮側偔偲傕侾僙僢僩傪帩偭偰偄傞偺偩偐傜丄RNA億儕儊儔乕僛偑嬫暿柍偔堚揱巕偺揮幨傪峴偊偽丄偳偺嵶朎傕傒側摨偠宍偱摨偠摥偒傪偡傞偙偲偵側傞丅偟偐偟幚嵺偼偦傫側偙偲偼側偄丅崱傑偱傕尒偰偒偨傛偆偵丄奺婍姱傪峔惉偟偰偄傞慻怐偼丄偳傟傕摨偠庴惛棏偵桼棃偡傞偺偵丄宍傕摥偒傕愮嵎枩暿偱偁傞丅偦傟偼側偤偩傠偆偐丅
丂偦偺棟桼偼丄偦傟偧傟偺嵶朎偱堚揱巕偺敪尰乮gene expression乯偑挷愡偝傟偰偄偰丄摿掕偺堚揱巕僒僽僙僢僩偟偐敪尰偟側偄偐傜偱偁傞丅
丂偦傟偱偼丄堚揱巕偺敪尰偼偳偺傛偆偵挷愡偝傟偰偄傞偺偩傠偆偐丅
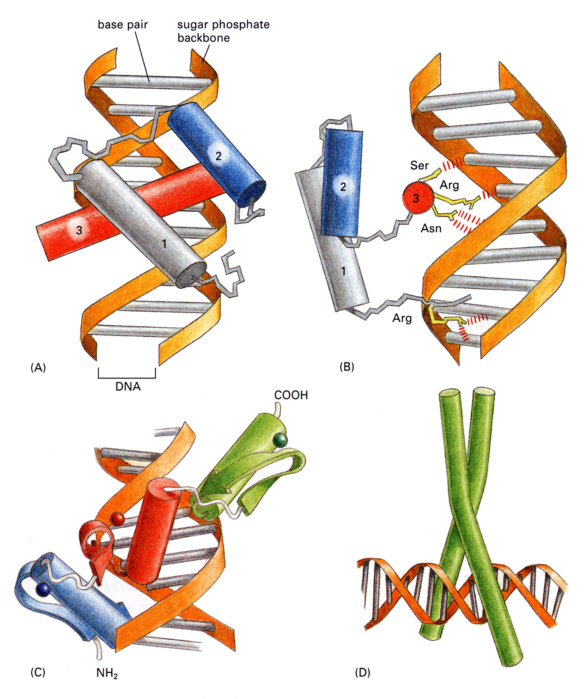
丂俢俶俙偺峔憿傪巚偄弌偟偰傎偟偄丅揮幨偟偨傝丄揮幨傪挷愡偡傞偨傔偵偼丄壗偐偑俢俶俙偵摥偒偐偗傞昁梫偑偁傞丅俢俶俙偵摥偒偐偗傞暘巕偼傗偼傝僞儞僷僋幙偱偁傞丅
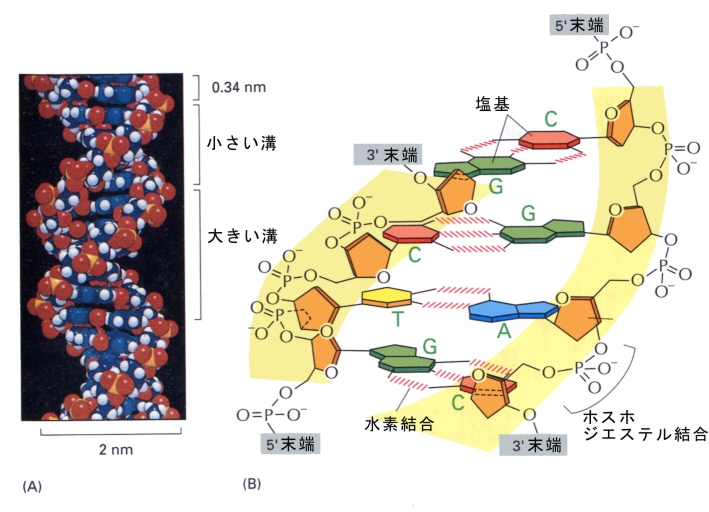
丂俢俶俙偺擇廳傜偣傫偵偼丄戝偒側峚乮major groove乯偲彫偝側峚乮minor groove乯偑岎屳偵偁傞丅偙偺峚偲丄憡曗揑側悈慺寢崌傪嶌偭偰偄側偄墫婎晹暘傪擣幆偟偰丄摿掕偺僞儞僷僋幙偑寢崌偡傞偙偲偑偱偒傞丅壓偺恾偼丄抐柺恾側偺偱擣幆晹埵傪1揰偩偗帵偟偰偁傞偑丄幚嵺偼偙偺傛偆側揰偑侾侽偐傜俀侽揰偁傝丄寢崌傪嫮偔偟偰偄傞丅
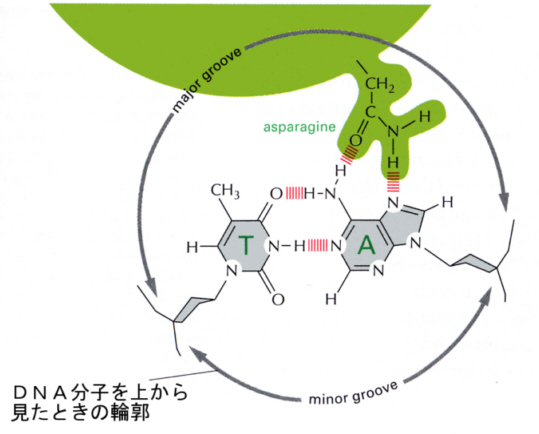
丂 俼俶俙億儕儊儔乕僛傗丄偙傟偐傜榖傪偡傞儗僾儗僢僒乕僞儞僷僋幙傕偙偺傛偆側寢崌傪嶌偭偰DNA偵摥偒偐偗偡傞丅
丂堦曽丄敪尰傪挷愡偡傞僞儞僷僋幙傕丄偙偺傛偆側寢崌傪嶌偭偰DNA偵摥偒偐偗傞丅壓偺恾偼丄敪尰挷愡僞儞僷僋幙偵嫟捠偵尒傜傟傞峔憿乮儌僠乕僼乯傪丄柾幃揑偵帵偟偨傕偺偱偁傞丅乮A乯偲乮B乯偼儂儊僆僪儊僀儞乮俀擭偱妛廗偡傞乯丄乮C乯偼僕儞僋僼傿儞僈乕乮zinc finger乯丄乮D乯偼儘僀僔儞僕僢僷乕偱偁傞丅
丂偝偰偙傟偩偗偺梊旛抦幆偱丄揮幨偑偳偺傛偆偵挷愡偝傟偰偄傞偐丄尨妀惗暔偲恀妀惗暔偱尒偰偄偔偙偲偵偟傛偆丅
侾亅侾丂堚揱巕偺峔憿
丂戝挵嬠乮Escherichia coli乯偺愼怓懱偼丄恀妀惗暔偺愼怓懱偺傛偆側暋嶨側峔憿偼偟偰偄側偄丅侾杮偺俢俶俙暘巕偑娐忬偵偮側偑偭偨扨弮側傕偺偱偁傞丅偙偺愼怓懱偼偦偺挿偝偐傜峫偊偰2000偐傜4000偺億儕儁僾僠僪嵔傪僐乕僪偟偰偄傞偲峫偊傜傟偰偄傞丅
丂夝摐偺奺抜奒偺峺慺傗丄俙俿俹偺惗嶻偵摥偔峺慺偼丄惗偒偰峴偔偨傔偵偼忢偵昁梫側偙偲偐傜丄揮幨偼偄偮傕婲偙偭偰偄傞丅偙偺傛偆側堚揱巕傪丄峔惉揑堚揱巕乮constitutive gene乯偲屇傫偱偄傞丅
丂俢俶俙偺堚揱忣曬傪倣俼俶俙偵揮幨偡傞偨傔偵偼丄偡偱偵弎傋偨傛偆偵俼俶俙億儕儊儔乕僛偑僾儘儌乕僞乕晹埵偵寢崌偟丄俢俶俙暘巕傪傎偳偒側偑傜丄揮幨奐巒晹埵偐傜廔椆晹埵傑偱丄俢俶俙偺拻宆嵔偺墫婎偵憡曗揑側僰僋儗僆僔僪嶰儕儞巁傪師乆偲屇傃崬傫偱丄寢崌傪怢偽偟偰峴偔昁梫偑偁傞丅
丂俼俶俙億儕儊儔乕僛偑寢崌偡傞僾儘儌乕僞乕晹埵偵偼丄堚揱巕偺庬椶偵偐偐傢傜偢丄憡摨側俀偮偺峔憿偑曐懚偝傟偰偄傞丅侾偮偼揮幨奐巒晹埵偐傜偍傛偦俇墫婎懳乮base pair, bp偲棯偡乯忋棳偺-10 region偲柤偯偗傜傟偨俇墫婎懳偱丄傕偆堦偮偼偝傜偵17bp忋棳偺-35 region 偲屇偽傟傞俇墫婎懳偺晹暘偱偁傞丅偙偺俀偮偺墫婎攝楍偵傛偭偰俼俶俙億儕儊儔乕僛偺寢崌偺巇曽偑寛傑傝丄偟偨偑偭偰偦偺恑傓曽岦丄偡側傢偪揮幨偺曽岦偑寛傑傞丅
丂RNA億儕儊儔乕僛偼丄壓偵帵偟偨揮幨廔寢晹埵傑偱偔傞偲僰僋儗僆僔僪嶰儕儞巁偺晅壛傪巭傔丄DNA偺拻宆嵔偲倣RNA傪棧偡丅
丂僐乕僪椞堟偼丄揮幨奐巒晹埵偲揮幨廔寢晹埵偵偼偝傑傟偰偄傞偙偲偵側傞丅
侾亅俀丂僆儁儘儞愢
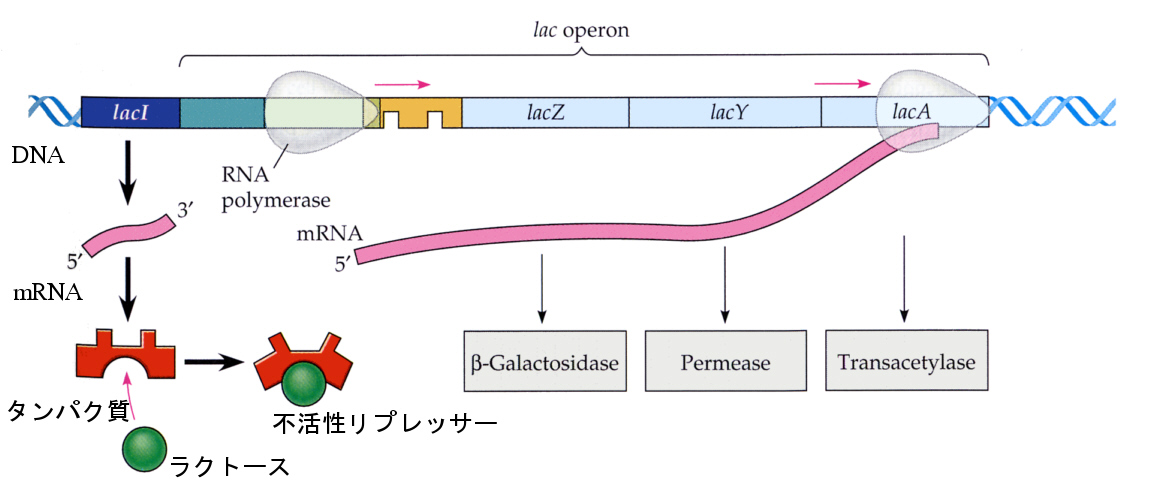
丂偝偰丄戝挵嬠傪僌儖僐乕僗偺偐傢傝偵儔僋僩乕僗乮僌儖僐乕僗偲僈儔僋僩乕僗偐傜側傞擇摐椶乯傪娷傓攟抧偱攟梴偡傞偲丄戝挵嬠偼傗偑偰儔僋僩乕僗傪夝摐偺弌敪暔幙偲偟偰巊偊傞傛偆偵側傞丅偙偺傛偆側忬嫷壓偱偼丄傆偩傫丄傎傫偺傢偢偐偟偐側偄丄儔僋僩乕僗傪僌儖僐乕僗偲僈儔僋僩乕僗偵暘夝偡傞兝僈儔僋僩僔僟乕僛丄儔僋僩乕僗傪嵶朎撪偵庢傝崬傓僷乕儈傾乕僛丄偦傟偲僩儔儞僗傾僙僠儔乕僛偺俁偮偺峺慺僞儞僷僋幙偑丄戝挵嬠嵶朎撪偵弌尰偟偰偔傞乮桿摫丄induction乯丅
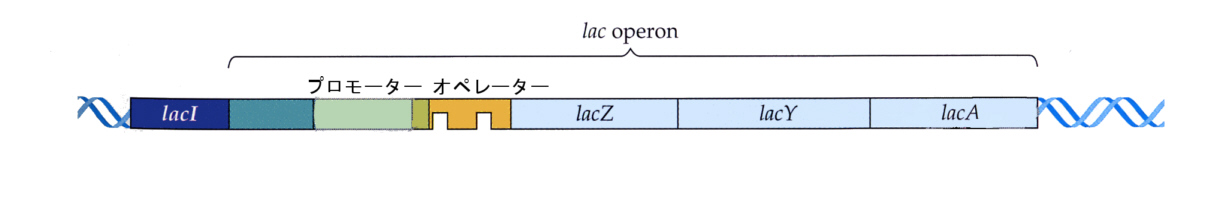
丂撍慠曄堎懱偺尋媶偐傜丄侾売強偺曄堎偱偙偺俁偮偺僞儞僷僋幙偑偡傋偰桿摫偝傟側偔側傞偙偲偑傢偐傝丄俁偮偺堚揱巕偑堦傑偲傑傝偵側偭偰偄偰丄傑偲傔偰敪尰偺挷愡偑偍偙側傢傟偰偄傞偲峫偊傜傟偨丅偙偺傛偆側暋悢偺堚揱巕偺傑偲傑傝偼僆儁儘儞乮operon乯偲柤偯偗傜傟偨丅
丂偦偺屻偺尋媶偵傛偭偰丄儔僋僩乕僗僆儁儘儞偼(lac僆儁儘儞)丄忋偵弎傋偨俁偮偺峺慺傪僐乕僪偡傞峔憿堚揱巕乮structural gene=protein-coding 乯偲丄偦偺偡偖忋棳偵偁傞僆儁儗乕僞乕晹埵傪娷傓僾儘儌乕僞乕晹埵偱峔惉偝傟偰偄傞偙偲偑傢偐偭偨丅俼俶俙億儕儊儔乕僛偼丄僾儘儌乕僞乕晹埵偵寢崌偟丄偦偺壓棳偵偁傞俁偮偺峔憿堚揱巕傪傑偲傔偰揮幨偡傞偨傔偵丄俁偮偺峺慺偼侾杮偺倣俼俶俙偐傜摨帪偵崌惉偝傟傞丅壓偺恾偱lacZ偼兝僈儔僋僔僟乕僛丄倢倎們倄偼僷乕儈傾乕僛丄lacA偼僩儔儞僗傾僙僠儔乕僛傪僐乕僪偡傞堚揱巕椞堟傪帵偡丅倢倎們俬偼師偵弎傋傞儕僾儗僢僒乕僞儞僷僋幙傪僐乕僪偡傞儕僾儗僢僒乕堚揱巕椞堟偱偁傞丅
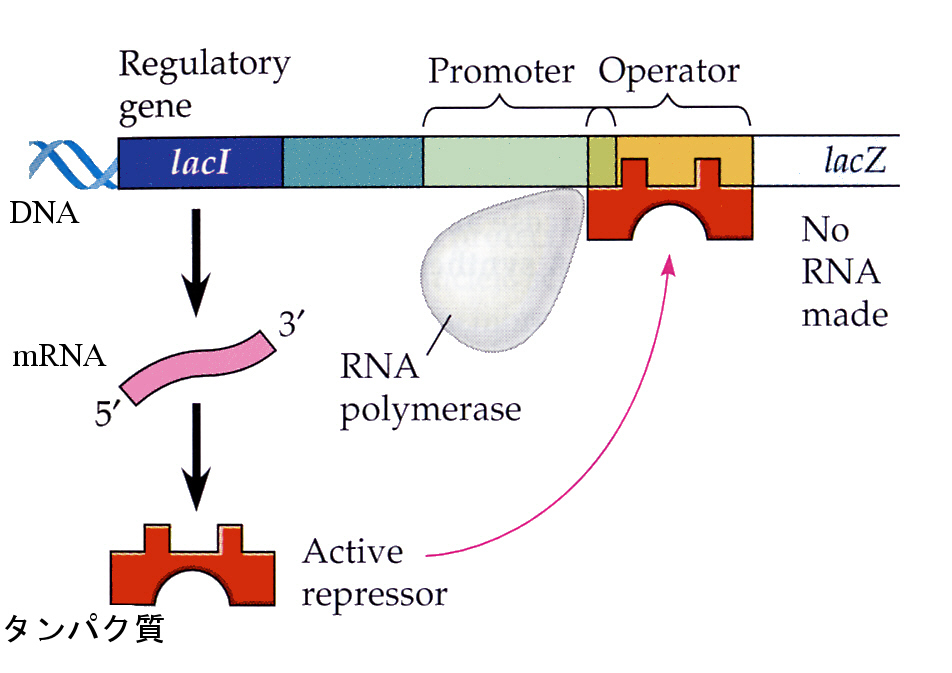
丂儔僋僩乕僗偺側偄娐嫬偱偼丄儔僋僩乕僗僆儁儘儞偺偝傜偵忋棳偵偁傞挷愡堚揱巕乮儕僾儗僢僒乕堚揱巕乯偺堚揱忣曬偐傜嶌傜傟傞儕僾儗僢僒乕僞儞僷僋幙偑忢偵崌惉偝傟偰偄偰丄僾儘儌乕僞晹埵撪偵偁傞僆儁儗乕僞乕晹埵偲嫮偔寢崌偡傞偨傔偵丄偙偺儕僾儗僢僒乕僞儞僷僋幙偑幾杺偵側偭偰丄僾儘儌乕僞乕晹埵偵俼俶俙億儕儊儔乕僛偑寢崌偱偒側偄丅偦偺偨傔偵忋婰偺俁偮偺僞儞僷僋幙偼崌惉偝傟側偄丅
丂儔僋僩乕僗偑攟抧偵懚嵼偡傞偲丄彮悢偺儔僋僩乕僗偑戝挵嬠撪偵擖偭偰戙幱偝傟丄偙傟偑儕僾儗僢僒乕僞儞僷僋幙偺傾儘僗僥儕僢僋寢崌晹埵偵寢崌偡傞丅偡傞偲丄儕僾儗僢僒乕僞儞僷僋幙偺峔憿偑曄傢傞偨傔丄僆儁儗乕僞乕晹埵偐傜儕僾儗僢僒乕僞儞僷僋幙偑奜傟傞丅偦偺偨傔丄僾儘儌乕僞乕晹埵偵俼俶俙億儕儊儔乕僛偑寢崌偱偒傞傛偆偵側傝丄倣俼俶俙偺揮幨偑巒傑傞丅偙偆偟偰丄儔僋僩乕僗傪夝摐偵棙梡偱偒傞傛偆偵側傞偺偱偁傞丅
丂偙偺曽幃偼丄偄偮傕偼僗僀僢僠偑僆僼偵側偭偰偍傝丄昁梫偵墳偠偰僗僀僢僠傪僆儞偵偡傞曽幃偱偁傞丅
http://esg-www.mit.edu:8001/esgbio/pge/lac.html
丂堦曽丄傾儈僲巁偺堦偮偱偁傞僩儕僾僩僼傽儞偼丄偄偔偮偐偺僗僥僢僾偱惗崌惉偝傟傞偑丄偦傟偧傟偺僗僥僢僾傪怗攠偡傞峺慺偼儔僋僩乕僗僆儁儘儞偺傛偆偵丄傂偲傑偲傔偺僆儁儘儞偲側偭偰偄傞丅
丂挷愡堚揱巕偺忣曬偐傜嶌傜傟傞儕僾儗僢僒乕偼丄儔僋僩乕僗儗僾儗僢僒乕偺応崌偲堘偄丄偙偺傑傑偱偼僆儁儗乕僞乕晹埵偵寢崌偱偒側偄丅偟偨偑偭偰丄僩儕僾僩僼傽儞僆儁儘儞偼偄偮傕壱摥偟偰偄偰丄崌惉峺慺孮偑嶌傜傟丄僩儕僾僩僼傽儞偑惗崌惉偝傟傞丅
丂僩儕僾僩僼傽儞偺嫙媼検偑廀梫検傪忋夞傞偲丄僩儕僾僩僼傽儞偑偩傇偮偔偺偱丄晄昁梫側惗崌惉傪巭傔偨曽偑柍懯偑側偄丅幚嵺偵僩儕僾僩僼傽儞偼儕僾儗僢僒乕偲寢崌偟丄偦偺宍傪曄偊傞丅偡傞偲偙偺儕僾儗僢僒乕僞儞僷僋幙亅僩儕僾僩僼傽儞暋崌懱偼丄僆儁儗乕僞晹埵偲寢崌偱偒傞傛偆偵側傝丄偦偺寢壥俼俶俙億儕儊儔乕僛偑僾儘儌乕僞乕晹埵偵寢崌偱偒側偔側傞偨傔偵揮幨偑巭傑傞丅
丂偙偺曽幃偼丄偄偮傕偼僗僀僢僠偑僆儞偵側偭偰偍傝丄昁梫偵墳偠偰僗僀僢僠傪僆僼偵偡傞曽幃偱偁傞丅
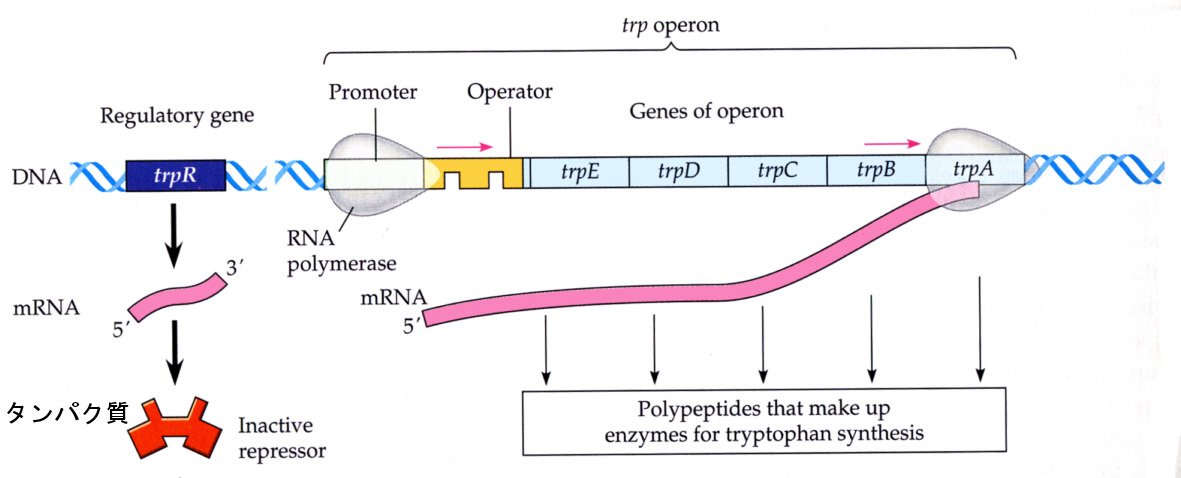
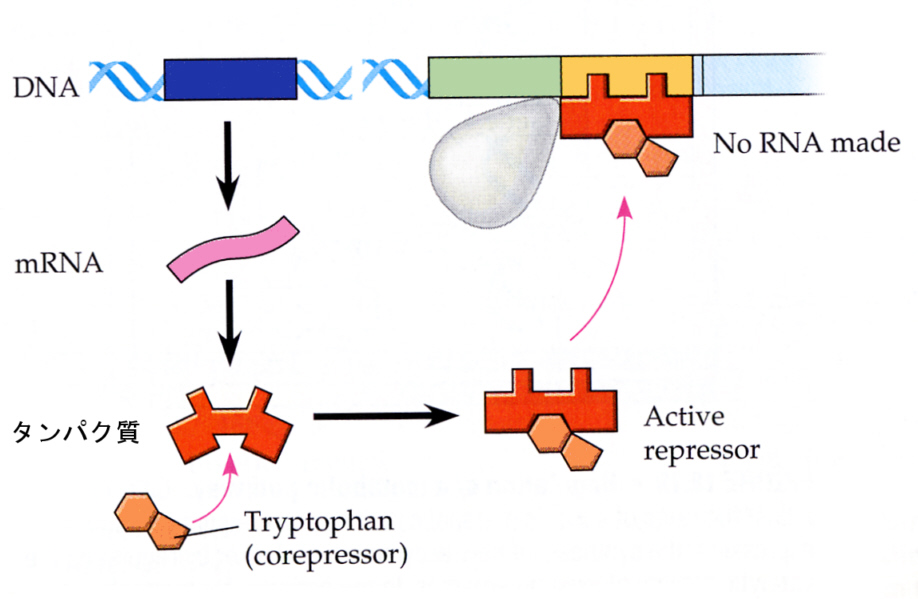
丂戝挵嬠偺尋媶偱傢偐偭偨偙偲偼丄敪尰偺挷愡偼偍傕偵揮幨偺儗儀儖偱峴傢傟偰偄傞偙偲丄傑偨愼怓懱偵偼僞儞僷僋幙傪僐乕僪偡傞椞堟埲奜偵丄忋偵弎傋偨傛偆側挷愡堚揱巕乮regulatory gene乯偑偁傞偙偲偱偁偭偨丅
丂恀妀惗暔偺愼怓懱偼丄尨妀惗暔偺傕偺偲斾傋傞偲偢偭偲暋嶨偱偁傞丅偟偨偑偭偰丄揮幨偲偦傟偵懕偔夁掱傕偢偄傇傫堎側偭偰偄傞丅堚揱巕偺敪尰偺挷愡偼丄揮幨儗儀儖偱偩偗偱側偔丄倣俼俶俙偺揮幨屻偵廋忺傗僾儘僙僢僔儞僌傪庴偗傞偙偲偵傛偭偰傕挷愡偝傟傞丅
丂恀妀惗暔偺応崌丄僾儘儌乕僞乕偐傜巒傑傞堚揱巕偵偼丄僞儞僷僋幙傪僐乕僪偟偰偄側偄椞堟偑娷傑傟偰偄傞丅僞儞僷僋幙傪僐乕僪偟偰偄傞椞堟傪僄僋僜儞乮exon乯偲偄偄丄僐乕僪偟偰偄側偄椞堟傪僀儞僩儘儞乮intron乯乮夘嵼攝楍丄intervening sequence偲傕乯偲尵偆丅
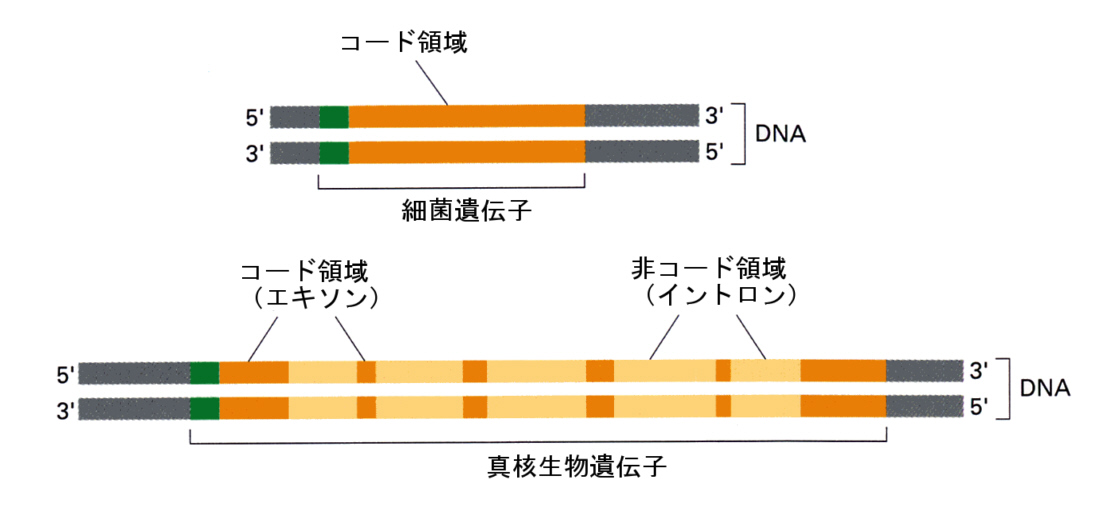
丂恀妀惗暔偺堚揱巕偺峔憿傪彮偟徻偟偔弎傋傛偆丅師偺墫婎攝楍偼僸僩兝亅僌儘價儞堚揱巕傪帵偟偰偄傞丅
乮Globin Gene Server傛傝乯
丂墫婎攝楍偼忋偐傜壓傊丄嵍偐傜塃傊暲傫偱偄偰丄嵟弶偺抜偺嵍抂偑俆乫懁偱偁傞丅俢俶俙擇杮嵔偺偆偪丄拻宆嵔偱偼側偄曽偺攝楍傪帵偟偰偁傞丅拻宆嵔傪巊偭偰倣俼俶俙傊揮幨偝傟傞偺偩偐傜丄嶌傜傟傞倣俼俶俙偼丄忋偺墫婎偺偆偪俿傪倀偵曄偊偨傕偺偱偁傞丅堚揱巕傪堦杮嵔偱帵偡偲偒偼偙偺傛偆側昞婰偺巇曽傪偡傞丅
丂侾乯忋俀抜偼僾儘儌乕僞乕椞堟偱丄俼俶俙億儕儊儔乕僛偑寢崌偟丄揮幨奐巒晹埵偐傜揮幨傪偼偠傔傞偨傔偵昁梫側椞堟偱偁傞丅俁偐強偺愒偄晹暘偼丄摿堎揑側僄儗儊儞僩偱丄摿偵嵟屻偺攝楍偼俿俙俿俙儃僢僋僗偲屇偽傟傞丅
丂俀乯悈怓偺晹暘偺愭摢偵偁傞俙俠俙俿俿TG偼揮幨奐巒揰偱丄僉儍僢僾攝楍偲傕屇偽傟傞丅偙傟偼偙偺晹暘偑廋忺傪庴偗偰僉儍僢僾峔憿偵側傞偐傜偱偁傞丅
丂俁乯嵁怓偺俙俿俧偑東栿奐巒僐僪儞偱偁傞丅揮幨奐巒晹暘偐傜東栿奐巒晹暘傑偱50墫婎懳棧傟偰偄傞偑(偙偺抣偼堚揱巕偵傛偭偰堎側傞乯丄儕乕僟乕攝楍偲屇偽傟傞丅
丂係乯巼怓偺晹暘偑僄僋僜儞偱丄崌寁俁偮偁傞丅嵟弶偺僄僋僜儞侾偼俋侽墫婎懳丄偡側傢偪俁侽傾儈僲巁傪僐乕僪偟偰偄傞丅
丂俆乯師偼僀儞僩儘儞侾偱丄侾俁侽墫婎懳偐傜側傝丄傾儈僲巁傪僐乕僪偟偰偄側偄椞堟偱偁傞丅
丂俇乯師偺巼怓晹暘偑僄僋僜儞俀偱丄俀俀俀墫婎懳偐傜側傝丄俁侾偐傜侾侽係傑偱偺俈係傾儈僲巁傪僐乕僪偟偰偄傞丅
丂俈乯俉俆侽墫婎懳偐傜側傞戝偒側僀儞僩儘儞俀偑師偵懕偔丅偙偺晹暘傕僌儘價儞僞儞僷僋幙偺峔憿偵偼慡偔娭學側偄丅
丂俉乯俁斣栚偺巼怓偑僄僋僜儞俁偱侾俀俇墫婎懳偐傜側傝丄侾侽俆偐傜侾係俇傑偱偺係俀傾儈僲巁傪僐乕僪偟偰偄傞丅
丂俋乯廔巒僐僪儞偼俿俙俙偱丄嵁怓偱帵偟偰偁傞丅
丂10乯俿俙俙偵懕偔悈怓偺晹暘偼丄俁乫懁偺揮幨偝傟傞偑東栿偝傟側偄晹暘偱偁傞丅偙偺晹暘偵偼愒怓偱俙俙俿俙俙俙偲帵偟偨攝楍傪娷傫偱偄傞偑丄偙偺晹暘偵俀侽侽偐傜俁侽侽偺億儕俙僥僀儖偑晅拝偡傞栚報偲側傞丅揮幨偝傟傞偲俙俙倀俙俙俙偲側傝丄偙偙偐傜偍傛偦俀侽墫婎懳壓棳偵億儕俙偑憓擖偝傟傞丅億儕俙僥僀儖偼丄倣俼俶俙偑暘夝偝傟傞偺傪慾巭偟丄庻柦傪挿偔偟偰偄傞丅
丂師偺恾偼丄堚揱巕偐傜丄倣俼俶俙偑揮幨偝傟傞條巕偺柾幃恾偱偁傞丅忋偺愢柧偲恾偐傜傢偐傞傛偆偵丄倣俼俶俙偼
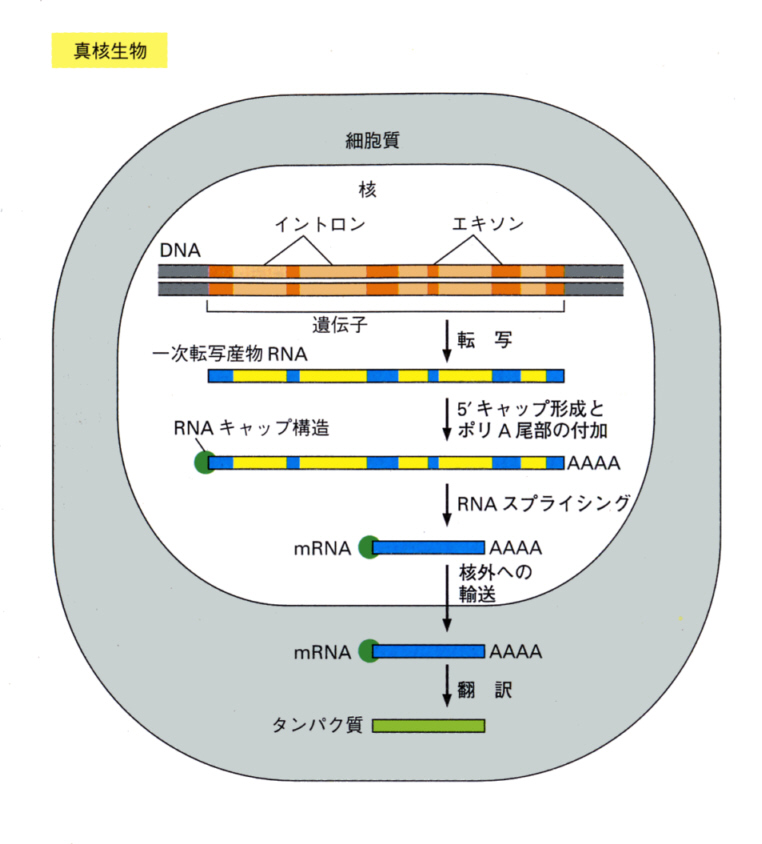
丂侾乯揮幨奐巒晹埵偐傜廔椆晹埵傑偱揮幨偝傟傞丅偙偺攝楍偵偼丄僄僋僜儞丄僀儞僩儘儞偲偲傕偵東栿偝傟側偄墫婎攝楍傪椉抂偵娷傓丅
丂俀乯俆乫懁偵俈亅儊僠儖僌傾僲僔儞乮Cap乯峔憿偑丄俁乫懁偵100亅250屄偺楢懕偟偨俙乮億儕俙僥僀儖乯偑晅壛偝傟傞丅
丂俁乯僀儞僩儘儞晹暘偑僗僾儔僀僔儞僌峺慺偵傛偭偰偮傑傫偱愗傜傟傞乮僗僾儔僀僔儞僌丄splicing乯丅
丂係乯姰惉偟偨倣俼俶俙乮惉弉倣俼俶俙乯偑妀枌岴偐傜僒僀僩僝乕儖傊弌傞丅
丂恀妀惗暔偺応崌偼丄尨妀惗暔偺僆儁儘儞偺傛偆側峔憿傪庢傜偢丄堦偮偺堚揱巕偼堦偮偺億儕儁僾僠僪嵔偟偐僐乕僪偟偰偄側偄丅偟偨偑偭偰忋偺恾偱昤偐傟偨堚揱巕撪偱偼丄侾偮偺億儕儁僾僠僪嵔偑俁偮偺僄僋僜儞偵暘抐偝傟偰攝抲偝傟偰偄傞偙偲偵側傞丅偳偆偟偰偙偆側偭偰偄傞偺偐偼傛偔夝偭偰偄側偄丅
丂恀妀惗暔偱傕丄僾儘儌乕僞乕椞堟偵俼俶俙億儕儊儔乕僛偑寢崌偟偰倣俼俶俙偺崌惉偑奐巒偝傟傞偲偄偆揰偼丄尨妀惗暔偲摨偠偱偁傞丅俼俶俙億儕儊儔乕僛偑擣幆偡傞墫婎攝楍偼丄撉傒巒傔偐傜30bp傎偳忋棳偵偁傞俿俙俿俙儃僢僋僗偲屇偽傟傞攝楍偱偁傞丅偝傜偵忋棳偵俉亅12bp偐傜側傞暋悢偺忋棳僾儘儌乕僞乕攝楍偑懚嵼偡傞丅
丂偝傜偵丄偢偭偲棧傟偨悢kbp忋棳偵偼僄儞僴儞僒乕偲屇偽傟傞椞堟偑偁偭偰丄偙偙偵傾僋僠儀乕僞乕僞儞僷僋幙偑寢崌偟丄揮幨偺懍搙傪崅傔偰偄傞丅
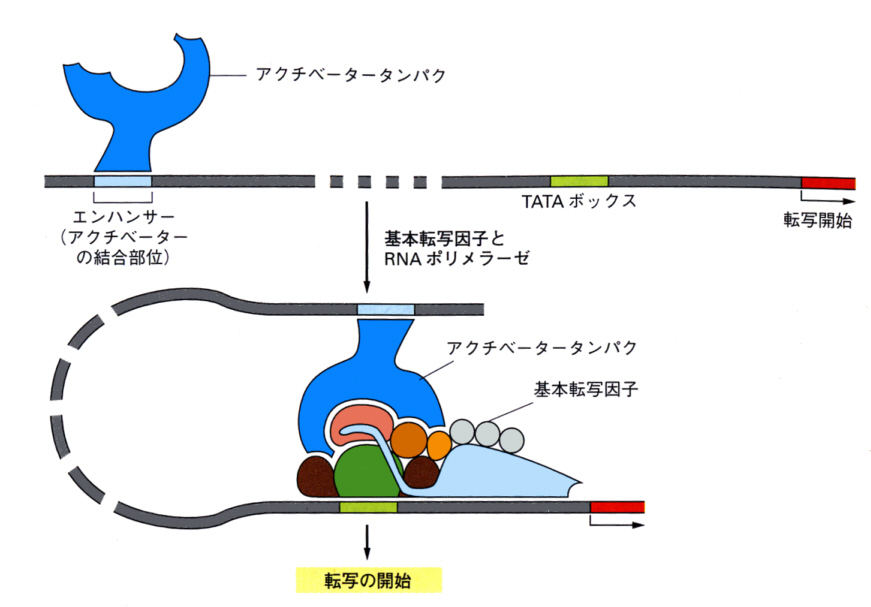
丂尨妀惗暔偺儕僾儗僢僒乕偺傛偆偵丄恀妀惗暔偱傕揮幨偺挷愡傪偡傞挷愡僞儞僷僋幙偑偨偔偝傫懚嵼偡傞丅偙傟傜偺僞儞僷僋幙偼俢俶俙偲寢崌偟偰丄揮幨偺奐巒丄廔椆丄揮幨検偺挷愡傪峴偭偰偄傞丅
丂僸僩偺僎僲儉俢俶俙検乮敿悢懱嵶朎丄偡側傢偪惛巕傗棏偁偨傝偺俢俶俙検乯偼丄戝挵嬠偺偍傛偦800攞偱丄3.2倶109偺墫婎懳傪娷傓偑丄堚揱巕偺悢偼俆枩偐傜10枩偔傜偄偱偁傠偆偲峫偊傜傟偰偄傞丅暯嬒300屄偺傾儈僲巁偐傜側傞僞儞僷僋幙傪嶌傞堚揱巕偼僐乕僪椞堟偩偗偱暯嬒300倶俁亖900墫婎懳昁梫偩偐傜丄10枩屄偺堚揱巕偺偨傔偵偼9倃107墫婎懳昁梫偲側傞丅偟偐偟偙傟偱傕慡僎僲儉偺俁亾偵偟偐偡偓側偄丅僀儞僩儘儞傗惂屼椞堟偑僐乕僪椞堟偺10攞偁偭偨偲偟偰傕丄偨偐偩偐30亾偵偟偐偡偓側偄丅偁偲偼梋暘側俢俶俙偲偄偆偙偲偵側傞乮忕挿丄redundancy乯丅
丂恀妀惗暔偺俢俶俙傪挷傋偰傒傞偲丄斀暅攝楍偲屇偽傟傞丄孞傝曉偟偑懡悢尒偮偐傞丅扨側傞僕儍儞僋側偺偩傠偆偲偄偆峫偊傗丄堚揱巕廳暋偱憹偊偰偟傑偭偨偑摿偵巟忈偑側偄偺偱曐懚偟偰偄傞偲偄偆峫偊丄壗偐摥偒偑偁傞偲偄偆峫偊側偳偑偁傝丄側偤偦偺傛偆側傕偺偑偁傞偐傛偔夝偭偰偄側偄丅傑偨丄偙偺側偐偵偼丄崱偼婡擻傪敪婗偟偰偄側偄偑丄婡擻揑側堚揱巕偺攝楍偲旕忢偵傛偔帡偨墫婎攝楍傪帩偭偨椞堟偑尒偮偐傞乮婾堚揱巕丄pseudogene乯丅
丂1970擭戙偺拞偛傠偐傜巒傑偭偨慻傒姺偊俢俶俙僥僋僯僢僋乮recombinant DNA technology乯乮偁傞偄偼墳梡媄弍傑偱傪娷傔偰堚揱巕岺妛genetic engineering偲傕尵偆乯偺敪揥偼丄偄傑傗惗暔妛偺尋媶偵寚偐偣側偄媄弍偺堦偮偵側偭偨丅偙偙偱偼偦偺曽朄偵偮偄偰奣娤偟偰傒傛偆丅
http://esg-www.mit.edu:8001/esgbio/rdna/rdnadir.html
丂僞儞僷僋幙偼傾儈僲巁偑堦偮側偑傝偵偮側偑偭偨傕偺偱偁傞乮堦師峔憿丄primary structure乯丅偟偨偑偭偰丄僞儞僷僋幙傪拪弌偟偰丄弮壔偟丄抂偐傜弴斣偵傾儈僲巁偺攝楍傪寛傔偰備偗偽丄僞儞僷僋幙偺堦師峔憿傪夝柧偡傞偙偲偑偱偒傞丅
丂偟偐偟側偑傜丄僞儞僷僋幙偼曄惈偟傗偡偐偭偨傝丄栚揑偲偡傞僞儞僷僋幙偑惗懱撪偱偼彮検偟偐懚嵼偟側偄偨傔偵丄拪弌偟偰弮壔偡傞偺偑擄偟偐偭偨傝偡傞応崌偑懡偄偺偱丄僞儞僷僋幙偐傜傾儈僲巁偺攝楍傪寛傔傞偺偼梕堈偱偼側偄丅
丂傾儈僲巁偺攝楍傪寛傔偰偄傞偺偼丄俢俶俙偺堚揱忣曬偱偁傝丄偦偙偐傜揮幨偝傟偨倣俼俶俙偺墫婎偺攝楍偱偁傞丅堚揱偺埫崋偑夝偭偨帪揰偱丄墫婎偺攝楍偑寛傑傟偽埫崋傪傕偲偵栠偟偰傾儈僲巁偺攝楍傪悇掕偱偒傞傛偆偵側偭偨丅
丂偦偙偱丄俢俶俙偺僞儞僷僋幙傪僐乕僪偟偰偄傞晹暘傪愗傝弌偟偨傝丄倣俼俶俙傪庢傝弌偟偰偦傟傪俢俶俙偵抲偒姺偊偰丄戝挵嬠側偳傪巊偭偰俢俶俙傪憹検偟丄墫婎偺攝楍傪寛傔傞曽朄偑奐敪偝傟偨丅
俀乯珲亖惂尷峺慺乮restriction enzyme乯偲屝亖俢俶俙儕僈乕僛
丂愼怓懱俢俶俙偼楢懕偟偨戝偒側暘巕偱偁傞丅偙傟傪傑傞偛偲庢傝埖偆偵偼戝偒偡偓傞丅僞儞僷僋幙偺傛偆偵揔摉側戝偒偝偺扨埵偺曽偑埖偄傗偡偄丅岾偄側偙偲偵丄俢俶俙忋偱偼丄挷愡椞堟傗僐乕僪椞堟偑晄楢懕乮旘傃旘傃偵懚嵼偡傞乯偱偁傞偙偲偑夝傝丄偆傑偔愗傝弌偣偽庢傝埖偄摼傞偙偲偑夝偭偨丅
丂偪傚偆偳偦偺崰丄墫婎偺攝楍偺摿掕偺応強偱俢俶俙傪愗抐偡傞峺慺偑嵶嬠偱尒偮偐偭偨丅壓偺恾偼Hind嘨偲偄偆惂尷峺慺偑丄5'-AAGCTT-3'偺夞暥峔憿傪偟偨墫婎攝楍傪擣幆偟偰丄灦宍偺埵抲偱俢俶俙傪愗抐偡傞柾幃恾偱偁傞丅
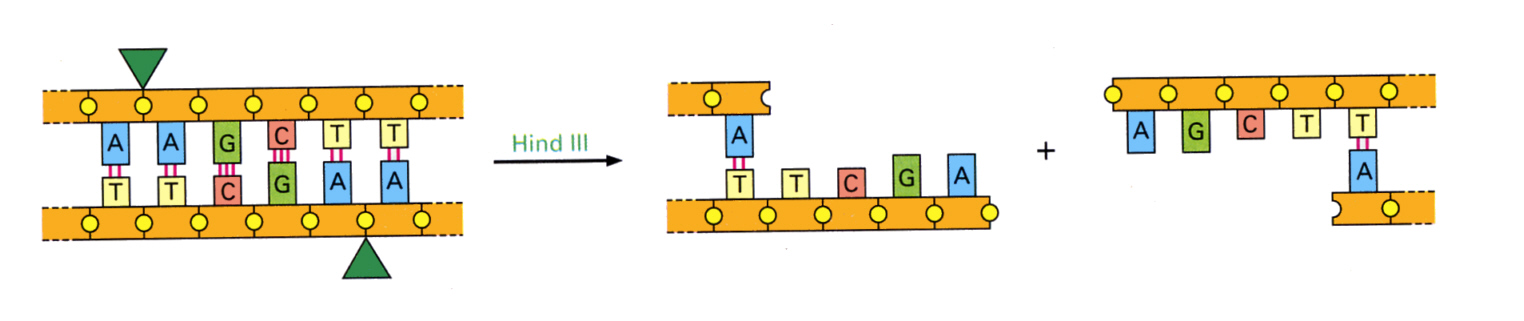
丂偙偺傛偆側愗抐偺巇曽傪偡傞偺偱丄椉抂偵屝戙偑偱偒傞偙偲偵側傞丅
丂忋偺恾偺愗抐偲斀懳偺偼偨傜偒傪偡傞偺偑丄DNA儕僈乕僛偱偁傞丅屝晅偗偟偨偄俢俶俙暘巕偺椉曽偵俆'-AAGCTT-3偲偄偆攝楍偑偁傟偽丄DNA儕僈乕僛傪巊偭偰DNA暘巕傪偮側偓崌傢偣傞偙偲偑偱偒傞丅
丂DNA暘巕拞偵暿偺DNA暘巕抐曅傪慻傒崬傓偨傔偵偼丄摨偠惂尷峺慺偱愗抐偟偰屝戙傪椉曽偺DNA偵偮偔傝丄俢俶俙儕僈乕僛偱屝晅偗偡傟偽傛偄乮慻傒姺偊乯丅
丂擣幆偡傞墫婎攝楍偺悢傗慻傒崌傢偣偺堎側傞惂尷峺慺偑丄懡悢敪尒偝傟偰偄傞丅暋悢偺惂尷峺慺傪巊偭偰愼怓懱俢俶俙傪愗抐偟丄揹婥塲摦憰抲偵偐偗偰戝偒偝偱暘偗丄惂尷峺慺愗抐売強偵傛傞俢俶俙抐曅偺攝楍抧恾傪嶌傞偙偲偑偱偒傞丅
丂偙偆偟偰丄DNA傪揔摉偺戝偒偝偵愗抐偟偨傝丄偮側偓崌傢偣偨傝偡傞偙偲偑偱偒傞傛偆偵側偭偨丅
丂偙偆偟偰丄愼怓懱俢俶俙偐傜揔摉側戝偒偝偺俢俶俙抐曅傪庤偵擖傟傞偙偲偑偱偒偨傜丄偙傟傪戝挵嬠偺僾儔僗儈僪偵丄忋偵弎傋偨珲偲屝傪巊偭偰慻傒崬傒丄戝挵嬠偵摫擖偡傞偙偲偑偱偒傞丅
丂僾儔僗儈僪偲偼丄廻庡愼怓懱偲偼撈棫偵懚嵼偟偰丄帺棩揑偵憹怋偟丄巕懛偵揱偊傜傟偰峴偔俢俶俙暘巕傪偄偆乮妀奜堚揱巕乯丅俢俶俙抐曅傪戝挵嬠撪偵塣傃崬傓丄偙偺傛偆側僾儔僗儈僪傪儀僋僞乕乮vector丄塣傃壆乯偲屇傇丅

丂栻嵻懴惈堚揱巕傪帩偭偨僾儔僗儈僪傪儀僋僞乕偲偟偰巊偄丄偦偺栻嵻傪娷傓攟抧偱戝挵嬠傪攟梴偡傟偽丄僾儔僗儈僪偑摫擖偝傟偨戝挵嬠偩偗偑惗偒巆傞丅
丂偙偆偟偰嶌惉偟偨戝挵嬠僋儘乕儞偺廤崌傪儔僀僽儔儕乕偲偄偆丅崱傑偱弎傋偨曽朄偱偼丄愼怓懱僎僲儉偐傜弌敪偟偰偄傞偐傜丄僎僲儉俢俶俙儔僀僽儔儕乕乮genomic DNA library乯偱偁傞丅
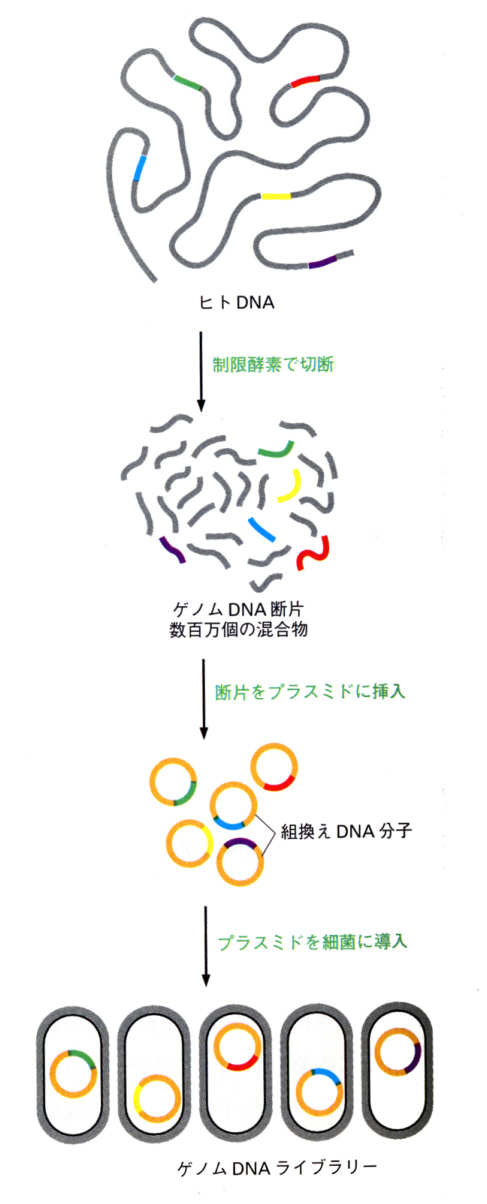
丂杮摉偵撉傒偨偄杮乮抦傝偨偄偲巚偭偰偄傞堚揱巕傪娷傓僋儘乕儞乯傪儔僀僽儔儕乕偐傜扵偡偨傔偵偼丄撪梕傪徠崌偡傞昁梫偑偁傞丅偦偺偨傔偵棙梡偝傟傞偺偑僾儘乕僽偱偁傞丅
丂婛抦偺僞儞僷僋幙偺堚揱巕傪扵偡偨傔偵偼丄偦偺傾儈僲巁攝楍偐傜倣俼俶俙偺墫婎偺攝楍傪悇掕偡傞丅側傞傋偔懳墳偡傞僐僪儞偺彮側偄傾儈僲巁乮儊僠僆僯儞傗僩儕僾僩僼傽儞傗僠儘僔儞側偳乯偑懕偔晹暘偑慖偽傟傞丅
丂偙偆偟偰丄恖岺揑偵憡曗揑俢俶俙傪崌惉偟丄曻幩惈摨埵尦慺偱昗幆偟偰僾儘乕僽傪嶌惉偡傞丅偙偺僾儘乕僽傪巊偊偽丄栚揑偺僞儞僷僋幙傪僐乕僪偡傞俢俶俙椞堟乮偁傜偐偠傔張棟傪巤偟堦杮嵔偵偡傞乯偲悈慺寢崌傪嶌傞偺偱丄栚揑偲偡傞俢俶俙傪摨掕偡傞偙偲偑偱偒傞丅
丂栚揑偲偡傞俢俶俙偺僾儘乕僽傪巊偊偽丄儔僀僽儔儕乕偺偳偺僋儘乕儞偵栚揑偺僞儞僷僋幙傪僐乕僪偡傞俢俶俙偑娷傑傟偰偄傞偐傪抦傞偙偲偑偱偒傞丅
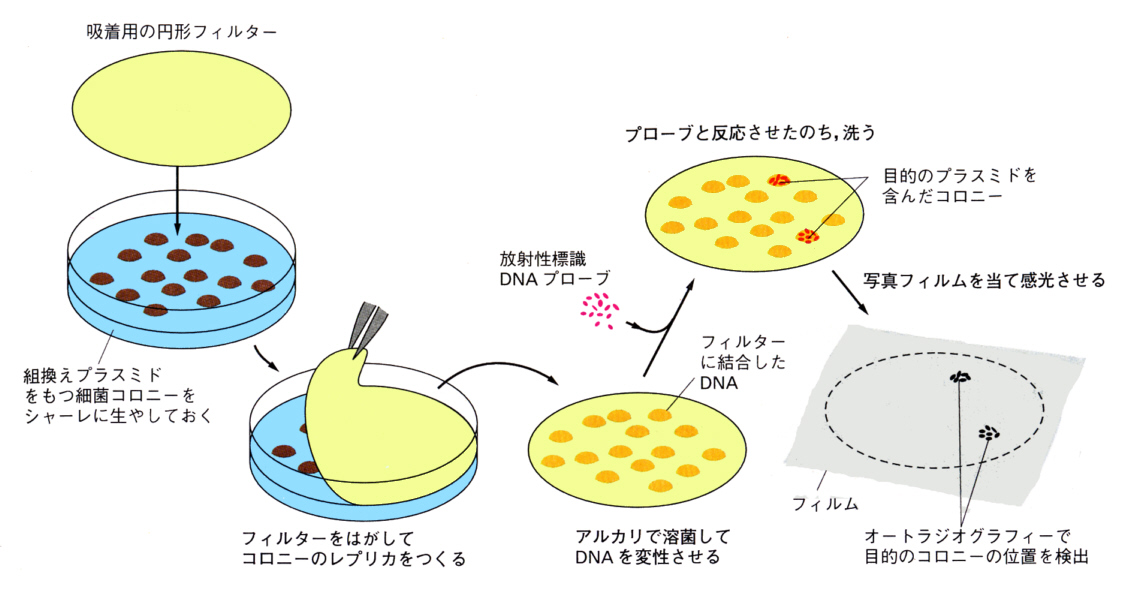
丂偙傟傑偱弎傋偨偺偼丄愼怓懱偐傜俢俶俙傪愗傝弌偟丄偦傟傪戝挵嬠傊摫擖偟丄栚揑偲偡傞俢俶俙抐曅傪娷傓僋儘乕儞傪扵偟弌偡曽朄偩偭偨丅
丂偟偐偟偙傟偼僔儑僢僩僈儞朄偲傕尵傢傟傞傛偆偵丄柍懯傕懡偄偟丄栚揑偺傕偺傪扵偟弌偡偺傕戝曄偱偁傞丅傕偆堦偮偺儔僀僽儔儕乕偺嶌傝曽偼丄倣俼俶俙偐傜嶌傞曽朄偱偁傞丅
丂摿掕偺憻婍傗敪惗偺摿掕偺帪婜偵丄摿掕偺僞儞僷僋幙偑懡検偵崌惉偝傟傞偙偲偑夝偭偰偄傟偽丄偦偙偐傜倣俼俶俙傪庢傝弌偟丄憡曗揑俢俶俙傪媡揮幨峺慺乮reverse transcriptase乯傪巊偭偰崌惉偡傞偙偲偑偱偒傞丅偙傟偼堦杮嵔側偺偱丄俢俶俙億儕儊儔乕僛傪巊偭偰偙傟傪擇杮嵔偵偟丄椉抂偵屝戙傪晅偗偰僾儔僗儈僪傊慻傒崬傓偙偲偑偱偒傞丅
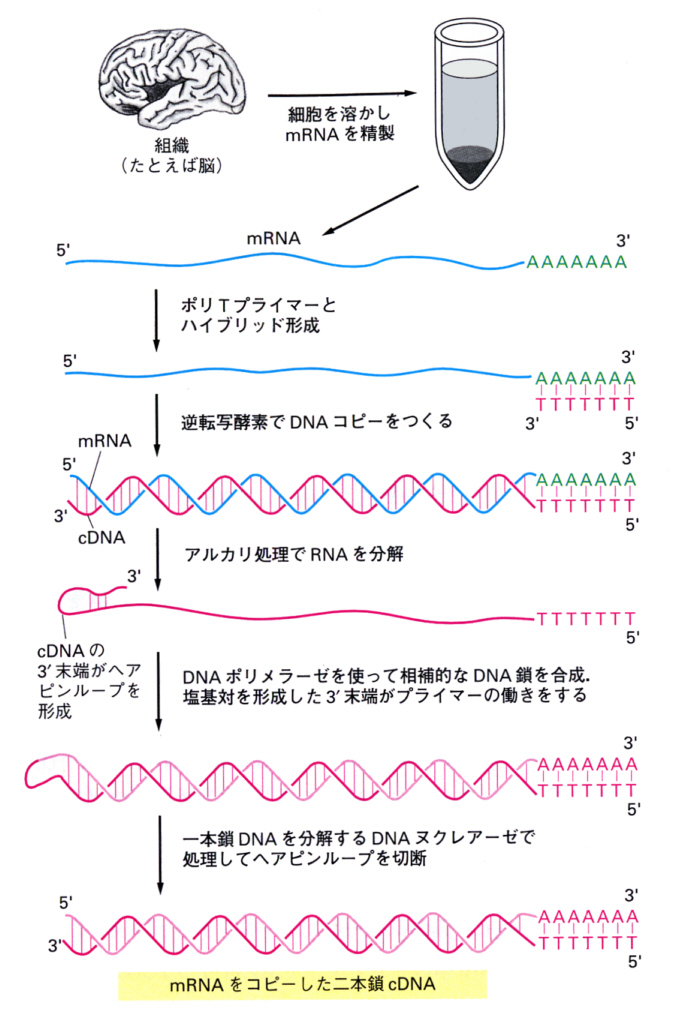
丂偁偲偼忋偵弎傋偨曽朄偲摨偠偱偁傞丅偙偆偟偰們俢俶俙儔僀僽儔儕乕乮cDNA library乯傪嶌傞偙偲偑偱偒傞丅
丂們俢俶俙儔僀僽儔儕乕偑僎僲儉俢俶俙儔僀僽儔儕乕偲戝偒偔堎側傞揰偼丄們DNA儔僀僽儔儕乕偑愼怓懱偺堚揱巕偺揮幨偝傟偨椞堟丄偟偐傕僄僋僜儞椞堟偺傒偺儔僀僽儔儕乕偱偁傞偙偲偱偁傞丅
丂偙偆偟偰丄栚揑偲偡傞俢俶俙傪娷傓戝挵嬠僋儘乕儞偑摿掕偱偒偨傜丄戝挵嬠傪攟梴偟偰戝検偵憹傗偟丄墫婎偺攝楍偺寛掕偵夞偡偙偲偑偱偒傞丅嵟嬤偱偼丄俹俠俼乮polymerase chain reaction乯朄傪巊偭偰丄彮検偺俢俶俙抐曅偐傜斾妑揑娙扨偵検傪憹傗偡偙偲偑偱偒傞傛偆偵側偭偨丅
丂墫婎攝楍傪寛掕偡傞曽朄偼偄偔偮偐奐敪偝傟偰偄傞偑丄偙偙偱偼嫵壢彂偵彂偐傟偨曽朄傪愢柧偡傞丅栚揑偲偡傞堦杮嵔偺俢俶俙抐曅偲俢俶俙億儕儊儔乕僛丄俢俶俙崌惉偺尨椏偲側傞倓俙俿俹丄倓俿俿俹丄倓俧俿俹丄倓俠俿俹丄偦傟偲儔僕僆傾僀僜僩乕僾乮俼俬乯偱昗幆偟偨倓倓俙俿俹傪僀儞僉儏儀乕僩偟偰丄俢俶俙崌惉傪峴偆丅偡傞偲崌惉偝傟傞俢俶俙偺嵔偺拞偵丄倓俙俿俹偺偐傢傝偵倓倓俙俿俹偑尨椏偲偟偰儔儞僟儉偵庢傝崬傑傟偰偟傑偄丄偦偙偱偦傟埲忋偺嵔偺怢挿偑巭傑偭偰偟傑偆丅
丂係偮偺墫婎偱偙偺憖嶌傪孞傝曉偟丄係偮暲傋偰億儕傾僋儕儖傾儈僪僎儖揹婥塲摦乮俹俙俧俤乯傪峴偆偲丄嵔偺抁偄傕偺傎偳尨揰偐傜墦偔傑偱塲摦偡傞丅係杮偺僇儔儉傪傑偲傔偰倃慄幨恀僼傿儖儉偵嵹偣偰姶岝偝偣尰憸偡傞偲丄俼俬昗幆倓倓僰僋儗僆僠僪偺偁傞偲偙傠偑僶儞僪偲偟偰尰傟偰偔傞丅
丂壓偐傜弴斣偵係偮偺僇儔儉偺僶儞僪傪偨偳偭偰峴偔偲丄偙偺暲傃曽偑尦偺俢俶俙偺墫婎偺攝楍偺3'仺5'偺憡曗揑攝楍偵側傞丅丂嵟屻偵丄惂尷峺慺偵傛傞抧恾傪傕偲偵丄尦偺堚揱巕偺慡懱憸傪嵞峔抸偡傞丅 丂墫婎偺攝楍偑寛掕偝傟傞偲丄堚揱巕僨乕僞僶儞僋偵搊榐偡傞丅偙偙偵偼悽奅拞偐傜廤傔傜傟偨堚揱巕偺墫婎攝楍偑搊榐偝傟偰偄偰丄僐儞僺儏乕僞僱僢僩儚乕僋傪夘偟偰徠崌偡傞偙偲偑偱偒傞丅帺暘偺尒偮偗偨堚揱巕偑偳偺堚揱巕偺墫婎攝楍偲帡偰偄傞偐側偳傕丄偡偖偵挷傋傞偙偲偑偱偒傞丅
俈乯堚揱巕摫擖摦暔乮transgenic organism乯
丂堚揱巕俢俶俙傪摦暔偵摫擖偡傞偙偲傕壜擻偲側偭偨丅儅僂僗偵惉挿儂儖儌儞堚揱巕傪摫擖偟丄僗乕僷乕儅僂僗傪嶌偭偨偲偄偆僯儏乕僗傪暦偄偨偙偲偑偁傞偩傠偆丅
http://www.ultranet.com/~jkimball/BiologyPages/R/RecombinantDNA.html